วิธีการติดฉลาก Digoxigenin (DIG
Section Overview
- การติดฉลาก Digoxigenin (DIG) และ Anti-dig Antibody
- การติดฉลาก DNA ด้วย PCR
- ขุดสุ่มพรีเมดดีเอ็นเอติดฉลาก
- Nick Translation Labeling ของ dsDNA สำหรับ โพรบในไซต์
- การติดฉลากแบบ Transcriptional ของ RNA Probes
- ขุด oligonucleotide 5 ' สิ้นสุดการติดฉลาก 3 ' สิ้นสุดการติดฉลากและ 3 ' หางการติดฉลาก
- การประมาณผลการผลิตของโพรบโดยกระบวนการตรวจจับโดยตรง
- ดาวน์โหลดและแหล่งข้อมูลที่เกี่ยวข้องกับการขุด
การติดฉลาก Digoxigenin (DIG) และ Anti-dig Antibody
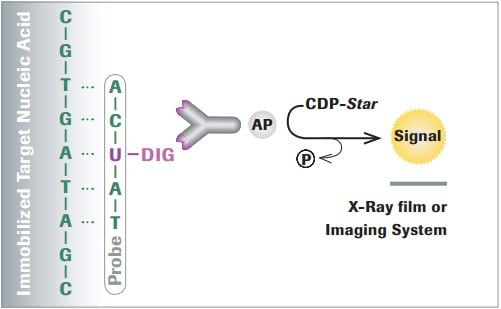
ขุดระบบเป็นเทคโนโลยี nonradioactive ที่เลือกที่จะติดฉลากและตรวจสอบกรดนิวคลีอิกสำหรับการใช้งานหลาย ระบบจะขึ้นอยู่กับเตียรอยด์ที่แยกจากพืช digitalis (Digitalis purpurea และ Digitalis lanata) พืชเหล่านี้เป็นแหล่งธรรมชาติเพียงแห่งเดียวของ digoxigenin ดังนั้นแอนติบอดีต่อต้านการขุดจึงไม่ได้ผูกติดกับวัสดุชีวภาพอื่นๆเพื่อให้มั่นใจว่ามีการติดฉลากเฉพาะ เนื่องจากความจำเพาะสูงนี้วัสดุที่น้อยกว่าจำเป็นเมื่อเทียบกับการติดฉลากกัมมันตรังสีทำให้ระบบขุดเหมาะสำหรับการวิเคราะห์การผสมของนิวเคลียส กรดนิวคลีอิก Immobilized จะถูกผสมกับโพรบที่ติดฉลากด้วยการขุดและการตรวจจับที่ตามมาจะดำเนินการโดยใช้ แอนติบอดี Anti-Digoxigenin ความสัมพันธ์สูงควบคู่ไปกับอัลคาไลน์ฟอสฟาเทส (AP) horseradish peroxidase (HRP) ฟลูออเรสเซีนหรือ rhodamine สำหรับการตรวจจับสีและสารเคมีหรือฟลูออเรสเซนต์

รูปที่ 1ตัวอย่างการตรวจจับกรดนิวคลีอิกที่ติดฉลากด้วยการใช้สารตั้งต้นเคมี
การติดฉลาก DNA ด้วย PCR
การติดฉลาก PCR เป็นวิธีที่ควรใช้ในการเตรียมโพรบที่ติดฉลากด้วยการขุดเมื่อเทมเพลตมีให้ใช้งานในปริมาณที่จำกัดเท่านั้นได้รับการทำให้บริสุทธิ์บางส่วนหรือสั้นมาก ซึ่งต้องใช้การปรับให้เหมาะสมน้อยกว่าวิธีการอื่นๆและให้ผลการผลิตสูงของโพรบที่ติดฉลากไว้ ในการติดฉลาก PCR พอลิเมอเรสแบบเทอร์โมสแตทจะรวม DIG dUTP เข้าด้วยกันเพื่อขยายขอบเขตเฉพาะของดีเอ็นเอแม่แบบ ผลลัพธ์ที่ได้คือโพรบการผสมข้ามพันธุ์ที่มีการติดฉลากสูงเฉพาะเจาะจงและมีความไวสูง
Reaction Principle
ในระหว่างปฏิกิริยา PCR มาตรฐาน Digoxigenin-11 dUTP จะถูกรวมเข้ากับ DNA ที่สังเคราะห์ขึ้นใหม่ สิ่งเดียวที่ต้องมีคือต้องมีข้อมูลลำดับบางอย่างของลำดับเป้าหมายเพื่อสังเคราะห์ไพรเมอร์ที่เหมาะสม ระบบการขุดที่ไม่มีกัมมันตภาพรังสีใช้ digoxigenin ซึ่งเป็นสเตียรอยด์ hapten เพื่อติดฉลาก DNA, RNA หรือ oligonucleotides สำหรับการผสมพันธุ์และการตรวจจับสีหรือเรืองแสงตามมา digoxigenin เป็นคู่กับ dUTP ผ่านพันธะเอสเทอร์อัลคาไล - เลไนล์ การสังเคราะห์กรดนิวคลีอิกเอนไซม์ที่ติดฉลากด้วยดีเอ็นเอพอลิเมอราสสามารถนำมารวมกันได้อย่างง่ายดาย การรวมกันของการติดฉลากที่ไม่ใช่กัมมันตรังสีกับ PCR เป็นเครื่องมือที่มีประสิทธิภาพสำหรับการวิเคราะห์ผลิตภัณฑ์ PCR และสำหรับการเตรียมตัวของการตรวจสอบฉลากจากลำดับเป้าหมายที่เกี่ยวข้องจำนวนเล็กน้อย
การติดฉลาก DNA ด้วยคุณสมบัติ PCR และประโยชน์
PCR Conditions
- เพิ่มประสิทธิภาพพารามิเตอร์การขยาย PCR (สภาพการขี่จักรยาน, ความเข้มข้นของแม่แบบ, ลำดับไพรเมอร์และความเข้มข้นของไพรเมอร์) สำหรับแต่ละแม่แบบและไพรเมอร์ที่ตั้งค่าไว้ในกรณีที่ไม่มี DIGDUTP ก่อนที่จะพยายามรวมตัวกันของการขุด
Template
- เพื่อให้ได้ผลลัพธ์ที่ดีที่สุดให้ใช้เม็ดมีดที่โคลนเป็นเทมเพลต ดีเอ็นเอจีโนมิกอาจใช้งานได้ยากขึ้น
- ความเข้มข้นของเทมเพลตมีความสำคัญต่อการผลิตโพรบเฉพาะอย่างที่ประสบความสำเร็จ
การติดฉลาก
PCR DIG Probe Synthesis Ki trequires น้อยกว่าวิธีการติดฉลากส่วนใหญ่เนื่องจากมี Expand High Fidelity Enzyme Blend ประโยชน์ของเอนไซม์ผสมนี้รวมถึง:
- สามารถใช้ภูมิภาคที่มีข้อมูลของ GC เป็นเทมเพลตได้อย่างมีประสิทธิภาพ
- สำหรับเทมเพลตส่วนใหญ่ไม่จำเป็นต้องมีการเพิ่มประสิทธิภาพของ MgCl 2 ปฏิกิริยาการเข้มข้นและการติดฉลากจะทำงานร่วมกับความเข้มข้นมาตรฐาน 1.5 มม. MgCl2
ขุดสุ่มพรีเมดดีเอ็นเอติดฉลาก
วิธีการติดฉลาก DNA แบบ " สุ่ม " นั้นขึ้นอยู่กับการผสมของ hexanucleotides ที่เป็นไปได้ทั้งหมดเข้ากับเทมเพลต DNA การผสมลำดับทั้งหมดจะแสดงในส่วนผสมของไพรเมอร์ hexanucleotide ซึ่งนำไปสู่การมีผลผูกพันของไพรเมอร์กับดีเอ็นเอแม่แบบในลักษณะทางสถิติ ดังนั้นจึงรับประกันระดับการติดฉลากที่เท่ากันตลอดความยาวทั้งหมดของดีเอ็นเอเทมเพลต สารเสริมถูกสังเคราะห์จาก 3 OH Termini ของไพรเมอร์ hexanucleotide แบบสุ่มโดยใช้เอนไซม์ Klenow เกรดการติดฉลาก ไตรฟอสเฟต deoxyribonucleoside ดัดแปลง ([2P] - [3S] 35 - [3H] 3 - [32 125 I] - digoxigenin หรือ biotinlabeled) ที่มีอยู่ในปฏิกิริยาจะรวมอยู่ในสายดีเอ็นเอเสริมที่สังเคราะห์ขึ้นใหม่
โพรบที่ติดฉลากเหล่านี้เหมาะอย่างยิ่งสำหรับการตรวจจับยีนสำเนาเดียวบน blots ใต้จีโนมิกการคัดกรองไลบรารี recombinant blots/slots และ blots ทางเหนือ เนื่องจากไพรเมอร์แต่ละตัวมีลำดับหกฐานที่แตกต่างกันผลิตภัณฑ์โพรบที่ติดฉลากจะเป็นคอลเลกชันของชิ้นส่วนของความยาวตัวแปร ดังนั้นโพรบที่ติดฉลากจะปรากฏเป็นรอยเปื้อนแทนที่จะเป็นแถบเฉพาะบนเจล การกระจายขนาดของโพรบที่ติดฉลากจะขึ้นอยู่กับความยาวของเทมเพลตต้นฉบับ
Reaction Principle
ในการติดฉลากเบื้องต้นแบบสุ่ม Klenow Enzyme จะคัดลอกเทมเพลต DNA ในที่ที่มีไพรเมอร์ Hexamer และ alkalilabile dig 11 dUTP โดยเฉลี่ยแล้วเอนไซม์จะแทรกความอิ่มตัวของการขุดหนึ่งครั้งในทุกๆการยืดตัวของนิวคลีโอไทด์ 20-25 ตัว ผลิตภัณฑ์ที่ติดฉลากที่ได้คือโพรบการผสมข้ามพันธุ์ที่ติดฉลากอย่างเป็นเนื้อเดียวกันซึ่งสามารถตรวจจับดีเอ็นเอเป้าหมายได้เพียง 0.10 – 0.03 น.
การติดฉลากการแปลของ Nick DNA สำหรับ ผู้ทดลองในไซต์
วิธีการแปล Nick ขึ้นอยู่กับความสามารถของ DNase I ในการแนะนำ nicks กระจายแบบสุ่มเข้าไปใน DNA ที่ความเข้มข้นของเอนไซม์ต่ำในการปรากฏตัวของ Mg2+ E. coli DNA polymerase ฉันสังเคราะห์ดีเอ็นเอที่เสริมเข้ากับเส้นที่สมบูรณ์ในทิศทาง 5 - 3 ' โดยใช้เทอร์มินิ 3 '-OH ของนิคเป็นไพรเมอร์ กิจกรรม exonucleolytic 5 - 3 ของ DNA Polymerase ฉันจะขจัดนิวคลีโอไทด์ในทิศทางของการสังเคราะห์ กิจกรรมพอลิเมอเรสตามลำดับจะแทนที่นิวคลีโอไทด์ที่ถูกลบด้วยไตรฟอสเฟต deoxyribonucleoside ที่ติดฉลากด้วยไอโซโทปหรือมีรูปร่าง ที่อุณหภูมิต่ำ (+15°C) ดีเอ็นเอที่ไม่มีฉลากในปฏิกิริยาจึงถูกแทนที่ด้วยดีเอ็นเอที่มีการสังเคราะห์ใหม่ ขั้นตอนการปรับให้เข้ากันได้ของ Fo Rin ความยาวของชิ้นส่วนที่ติดฉลากซึ่งได้รับจากขั้นตอนนี้ควรอยู่ที่ประมาณ 200-500 ฐาน
การติดฉลากแบบ Transcriptional ของ RNA Probes
สำหรับบางแอปพลิเคชัน RNA ที่มีป้ายกำกับแบบดิจิตอลเป็นโพรบการผสมข้ามพันธุ์ที่มีประสิทธิภาพมากกว่าดีเอ็นเอที่มีป้ายกำกับแบบดิจิตอล ตัวอย่างเช่นโพรบ RNA ที่ติดฉลากด้วยการขุดจะสามารถตรวจจับ mRNA ที่หายากในปริมาณนาโนแกรมของ RNA ทั้งหมด โพรบ RNA ที่มีป้ายกำกับเหล่านี้จะถูกสร้างขึ้น จากเทมเพลต DNA ในวิธีการถอดสคริปต์ RNA ดีเอ็นเอจะถูกโคลนเข้าไปในไซต์การโคลนหลายไซต์ของเวกเตอร์การถอดสคริปต์ระหว่างโปรโมเตอร์สำหรับพอลิเมอไรเซส RNA ที่แตกต่างกัน (เช่น T7, SP6 หรือ T3 RNA polymerase) เทมเพลตจะถูกปรับให้เป็นเส้นตรงโดยการแยกตัวของเวกเตอร์ที่ไซต์ที่ไม่ซ้ำกัน (ใกล้กับเม็ดมีด) โพลิเมอร์ RNA จะถอด DNA ที่แทรกลงในสำเนา RNA ที่ต่อต้านการรับรู้เมื่อมีส่วนผสมของ ribonucleotides (รวมถึงขุด -UTP) ในระหว่างการ μg ปฏิกิริยาดีเอ็นเอสามารถถอดความได้หลายครั้ง (สูงสุดหนึ่งร้อยเท่า) เพื่อสร้างสำเนาอาร์เอ็น μg เอแบบเต็มความยาวขนาดใหญ่ที่ติดฉลากด้วยการขุดเจาะ (อาร์เอ็นเอ 10-20 μ m จากดีเอ็นเอ 1 μ m ในปฏิกิริยามาตรฐาน) ขุดจะรวมอยู่ในอาร์เอ็นเอที่ประมาณทุก 25-30 นิวคลีโอไทด์
Reaction Principle
เทมเพลต DNA ที่จะถอดสคริปต์จะถูกโคลนเข้าไปในเว็บไซต์ตัวเชื่อมโยงหลายตัวของเวกเตอร์การถอดสคริปต์ที่เหมาะสมซึ่งมีโปรโมเตอร์สำหรับโพลิเมอร์ SP6 และหรือ T3 และ T7 RNA หลังจากการปรับให้เป็นเส้นตรงที่ไซต์ที่เหมาะสมแล้ว RNA จะถูกถอดความในบริเวณที่มีการขุด 11 UTP ภายใต้สภาวะมาตรฐาน RNA ที่ติดป้าย μg กับการขุดแบบเต็มความยาวประมาณ 10 μ m จะได้รับการถอดป้าย μg จากเทมเพลต 1 μ m
เคล็ดลับต่อไปนี้เป็นสิ่งสำคัญสำหรับการติดฉลากโพรบ RNA ที่ประสบความสำเร็จ:
RNases
RNases มีอยู่ทั่วไปและไม่จำเป็นต้องใช้ปัจจัยร่วมในการทำกิจกรรม หากคุณต้องการประสบความสำเร็จให้ใช้ความระมัดระวังที่เป็นไปได้ทั้งหมดเพื่อป้องกันการปนเปื้อน RNase For instance:
- ขอแนะนำให้ใช้พลาสติกแบบใช้แล้วทิ้งเครื่องแก้วอบด้วยเตาอบหรือพลาสติกที่ผ่านการขจัดสิ่งปนเปื้อนด้วย RNase ZAP หรือน้ำยาที่คล้ายกัน
- เตรียมสารละลายทั้งหมดด้วยน้ำที่ผ่านการบำบัดด้วยไดเอทิลไพโรคาร์บอเนต (DEPC) หรือไดเมทิลไดคาร์บอเนต (DMDC) และนึ่งสารละลาย
- สวมถุงมือตลอดขั้นตอน
- ประสิทธิภาพการติดฉลากขึ้นอยู่กับความบริสุทธิ์ของแม่แบบดีเอ็นเอ เทมเพลตควรได้รับการทำให้บริสุทธิ์สูง
- เทมเพลตสุดท้ายต้องเป็นแบบลิเนียริไลซ์, ฟีนอล/คลอโรฟอร์มที่สกัดและเอทานอลตกตะกอน
Template Sequence
- ภูมิภาคไพรเมอร์และ/หรือโพลีลินเกอร์บางแห่งในเทมเพลต DNA นั้นเป็นที่ยอมรับในบางส่วนของลำดับ RNA ของ ribosomal 28 และ 18 วินาที ดังนั้นโพรบที่ติดฉลากอาจสร้างสัญญาณเฉพาะแต่ไม่ต้องการในตัวอย่างที่มี RNA ที่เด่นชัดเหล่านี้ เมื่อต้องการลดผลกระทบนี้ให้เอาลำดับของตัวเชื่อมโยงหลายตัวออกจากแม่แบบของคุณให้มากที่สุดเท่าที่จะทำได้
- หากคุณใช้ PCR เพื่อสร้างแม่แบบ DNA ผลิตภัณฑ์ของปฏิกิริยา Expand High Fidelity จะมีชิ้นส่วนบางส่วนที่มีระยะยื่น 3 นิ้ว ส่วนที่ยื่นออกมานี้อาจทำให้เกิดการห่อหุ้มผลิตภัณฑ์ในปฏิกิริยาการติดฉลากแบบ Transcriptional
Template Length
- ความยาวเทมเพลตที่เหมาะสมคือประมาณ 1 kb
- Minimum length should be at least 200 bp
การจัดเก็บโพรบ
- เพื่อความเสถียรในระยะยาวควรแยกส่วนโพรบ RNA และเก็บไว้ที่ -20°C หรือ -70°C
- โพรบ RNA ที่ติดฉลากด้วยการขุดจะมีความเสถียรอย่างน้อย 1 ปีที่อุณหภูมิ -20°C หรือ -70°C ในเอทานอล
Probe Sensitivity
- ในการตรวจสอบความไวของโพรบ Antisense RNA ที่ติดฉลากด้วย การขุดอย่างรวดเร็วให้เตรียม Sense RNA (ที่ไม่มีฉลาก) ที่สอดคล้องกันด้วยการถอดสคริปต์ จากนั้นใช้ทรานสคริปท์การรับรู้บริสุทธิ์ที่ความเข้มข้นที่แตกต่างกันเป็นเป้าหมายในเขตภาคเหนือ จากผลของการเกิด blot คุณสามารถกำหนดจำนวนเป้าหมายที่ต่ำที่สุดได้อย่างง่ายดาย (บันทึกการรับรู้) ซึ่งสามารถตรวจจับได้โดยการติดฉลาก probe (ทรานสคริปท์ antisense)
ขุด oligonucleotide 5 ' สิ้นสุดการติดฉลาก 3 ' สิ้นสุดการติดฉลากและ 3 ' หางการติดฉลาก
สำหรับการใช้งานบางอย่างเช่น การปรับให้เป็นซินซิทซิทยูไฮไดไรเซชันโพรบสังเคราะห์ที่ติดฉลากด้วยการขุดเป็นโพรบการผสมข้ามพันธุ์ที่ดีที่สุด นอกเหนือจาก การปรับแต่งการปรับแต่งการไหลเวียนของเลือดแล้วอาจใช้ oligonucleotides ที่มีป้ายกำกับการขุดเป็นโพรบการไหลเวียนของเลือดในการใช้งานจำนวนมากรวมถึงจุด/ช่องการคัดกรองไลบรารีการตรวจหาลำดับยีนซ้ำๆใน blots ภาคใต้และการตรวจหา mRNA ที่อุดมสมบูรณ์ใน blots ทางตอนเหนือ
มีหลายวิธีสำหรับการติดฉลากขุดของ oligonucleotides และสรุปไว้ด้านล่าง
ป้ายชื่อปลาย 5 ของ Oligonucleotide กับ DIG-NHS-Ester
การติดฉลากปลาย 3 องศาด้วยขุด - ddutp
เพิ่มหาง 3 ของ dig dutp และ datp (ประมาณ 40 - 50 ตกค้าง)

รูปที่ 2การตรวจสอบและการตรวจสอบ oligonucleotide ที่ไม่ใช่กัมมันตรังสี
การประมาณผลการผลิตของโพรบโดยกระบวนการตรวจจับโดยตรง
ในการเพิ่มโพรบในปริมาณที่ถูกต้องลงในการผสมข้ามพันธุ์คุณต้องกำหนดปริมาณโพรบที่ติดฉลากด้วยการขุดที่ผลิตในปฏิกิริยาการติดฉลากก่อน ขั้นตอนการตรวจจับโดยตรงที่ระบุไว้ในที่นี้จะเปรียบเทียบปริมาณของป้ายกำกับการขุดในชุดของการเจือจางที่เตรียมจากโพรบที่ติดฉลากด้วยความเข้มข้นที่ทราบของกรดนิวคลีอิกควบคุมที่ติดฉลากด้วยการขุด
หมายเหตุ: หากคุณติดฉลาก DNA probe โดย PCR คุณไม่จำเป็นต้องทำการตรวจจับโดยตรงเพื่อประเมินผลผลิต
สำหรับโพรบที่ติดฉลาก PCR ให้ใช้วิธีการประเมินอิเล็กโตรโฟเรซิสแบบเจล
การตรวจจับโดยตรงจะเกี่ยวข้องกับขั้นตอนต่อไปนี้:
- การเตรียมการเจือจางแบบอนุกรมของโพรบที่ติดฉลากไว้และการจำแนกบนเมมเบรนไนลอน (เวลาที่ต้องใช้: 15 นาที)
- การตรวจจับการขุดในจุดที่มีการเรืองแสงทางเคมี ; เวลาที่ต้องใช้ (2 ชั่วโมง)
ดาวน์โหลดและแหล่งข้อมูลที่เกี่ยวข้องกับการขุด
Need help? ดู คู่มือการเลือกผลิตภัณฑ์ขุดของเรา
For more information, view the following brochures:
สามารถดูข้อมูลทางเทคนิคโดยละเอียดได้ในคู่มือขุด:
- คู่มือแอปพลิเคชันขุดสำหรับการผสมตัวกรอง
- คู่มือการประยุกต์ใช้งานขุดสำหรับ Nonradiactive ใน การผสมซิตูฉบับที่ 4
เรียนรู้วิธีการจัดการระบบขุดในเคล็ดลับทางเทคนิคของเรา:
สำหรับการวิจัยวิทยาศาสตร์เพื่อชีวิตเท่านั้น Not for use in diagnostic procedures.
สินค้าที่เกี่ยวข้อง
เพื่ออ่านต่อ โปรดเข้าสู่ระบบหรือสร้างบัญชีใหม่
ยังไม่มีบัญชีใช่หรือไม่?