Buccal DNA Extraction และ WGA Amplification Protocol
โปรโตคอลนี้เป็นวิธีที่ง่ายและสะดวกในการแยกขยายและทำให้ดีเอ็นเอของยีโนมบริสุทธิ์จากเซลล์วัว การเก็บตัวอย่าง DNA เป็นวิธีที่สะดวก เมื่อดีเอ็นเอถูกแยกออกโดยใช้โปรโตคอลการสกัด miniprep มันสามารถขยายได้โดยใช้โปรโตคอลการขยายจีโนมทั้งหมด
โปรโตคอล Miniprep DNA จีโนมิก
เพื่อผลลัพธ์ที่ดีที่สุดให้ใช้ GenElute Mammalian Genomic DNA Miniprep Kit(G1N10)สำหรับกระบวนการนี้
- เช็ดก้านที่เก็บไว้ให้แห้งที่อุณหภูมิห้องเป็นเวลา 15 นาที
- เติมสารละลาย Lysis Solution T 280 μ L และ Proteinase K 20 μ m μL ใส่ก้านไม้แล้วค่อยๆหมุน μL ปิดฝาหลอดและผสมโดยการวน
- บ่มตัวอย่างที่อุณหภูมิ 55°C เป็นเวลา 20 นาทีโดยใช้น้ำวนเป็นครั้งคราว
- เติมสารละลาย Lysis 200 ไม μL รลิตร C และ Vortex ให้ทั่วเป็นเวลา 15 วินาที
- บ่มเชื้อที่อุณหภูมิ 70°C เป็นเวลา 10 นาที
- เติมสารละลายเตรียมคอลัมน์ 500 มล. ลงในคอลัมน์เย็บเล่ม GenElute Miniprep แต่ละคอลัมน์ (โอริงสีแดง) และ μL ไปปั่นเหวี่ยงที่แรงเหวี่ยง 12,000 × g เป็นเวลา 1 นาที ทิ้งของเหลวที่ไหลผ่าน
โซลูชันการเตรียมคอลัมน์ช่วยเพิ่มการยึดเกาะดีเอ็นเอให้กับเมมเบรนทำให้ได้ผลผลิตที่สม่ำเสมอมากขึ้น - เติมเอทานอล 200 ไม μL รลิตร 95 - 100% ลงในไลเซทจากขั้นตอนที่ 5
- ผสมให้เข้ากันโดยการวนและเพิ่มเนื้อหาทั้งหมดของหลอดลงในคอลัมน์ที่มีผลผูกพัน
- ปั่นเหวี่ยงที่ ≥6500 × g เป็นเวลา 1 นาที
- ทิ้งหลอดเก็บตัวอย่างที่มีการไหลผ่านและใส่แกนเข้าเล่มลงในหลอดเก็บตัวอย่างขนาด 2 มล. หลอดใหม่
- เติมน้ำยา μL ความสะอาด 500 มล. (อย่าลืมเจือจางด้วยเอทานอลก่อนใช้งานครั้งแรก) และปั่นเหวี่ยงที่แรงเหวี่ยง ≥6500 × g เป็นเวลา 1 นาที
- ทิ้งหลอดเก็บตัวอย่างและการไหลผ่านและใส่แกนที่เย็บเล่มลงในหลอดเก็บตัวอย่างขนาด 2 มล. ใหม่
- เติมน้ำยา μL ความสะอาด 500 000 3 มล. ลงในคอลัมน์ที่มีผลผูกพันและปั่นเหวี่ยงด้วยความเร็วสูงสุด (12,01-16,000 × g) เป็นเวลา 12 นาทีเพื่อทำให้คอลัมน์ที่มีผลผูกพันแห้ง
- ปิเปตต์ 200 ไมโครลิตรของสารละลายเจือจางลงบนแกน ≥รับการจับและปั่นเหวี่ยงเป็นเวลา 1 นาทีที่ μL 6500 × g
- เก็บดีเอ็นเอที่เจือจางที่อุณหภูมิ -20°C หรือไปยังขั้นตอนการขยาย
หากใช้ WGA 2 ไม่จำเป็นต้องจัดหา DNA polymerase เนื่องจากมีเอนไซม์มาพร้อมกับชุด
Whole Genome Amplification (WGA) Protocol
โปรโตคอลสำหรับ GenomePlex ทั้ง Genome Amplification ดำเนินการกับ GenomePlex ทั้ง Genome Amplification Kit (WGA1)และ/หรือชุดขยาย Genome ทั้งหมดที่สมบูรณ์ (WGA2)
การกระจายตัว
- เตรียมสารละลาย DNA ขนาด 1 นาโนกรัม/มล. จากโปรโตคอลการแยกเลือดครบตามที่อธิบายไว้ข้างต้น
- เติม µL บัฟเฟอร์ 1 แตกตัว 10 μ m µL 10 เท่าลงใน DNA ขนาด 1 μ m (µL นาโนกรัม/ตารางเมตร) ในหลอด PCR
- วางท่อในช่องระบายความร้อนที่อุณหภูมิ 95°C เป็นเวลา 4 นาที หมายเหตุการบ่มเชื้อต้องใช้เวลานานและการเบี่ยงเบนใดๆอาจเปลี่ยนแปลงผลลัพธ์ได้
- ทำให้ตัวอย่างเย็นลงทันทีบนน้ำแข็งและปั่นเหวี่ยงเป็นเวลาสั้นๆ
Library Preparation
- เพิ่ม µL บัฟเฟอร์ 2 รับเตรียมไลบรารี 1 หรือ ชุด
- เพิ่ม 1 ชุดของโซลูชันการ µL ให้ห้องสมุดเสถียร
- ผสมให้เข้ากันและใส่ในช่องระบายความร้อนที่อุณหภูมิ 95°C เป็นเวลา 2 นาที
- ทำให้ตัวอย่างเย็นลงบนน้ำแข็งและปั่นเหวี่ยงเป็นเวลาสั้นๆ
- เติมเอนไซม์เตรียมห้องสมุด 1 µL ผสมให้เข้ากันและปั่นเหวี่ยงเป็นเวลาสั้นๆ
- ใส่ตัวอย่างลงในไซโคลนความร้อนและบ่มดังนี้:
16°C เป็นเวลา 20 นาที
24°C เป็นเวลา 20 นาที
37°C เป็นเวลา 20 นาที
75°C เป็นเวลา 5 นาที
4°C - นำตัวอย่างออกจากเครื่องปั่นแบบใช้ความร้อนและปั่นเหวี่ยงเป็นเวลาสั้นๆ สามารถขยายตัวอย่างได้ทันทีหรือเก็บไว้ที่อุณหภูมิ - 20°C สูงสุดสามวัน
การขยายเสียง
- เพิ่มสารตัวกระ µL ต่อไปนี้ลงในปฏิกิริยา 15 μ s ทั้งหมด:
7.5 µL 10 เท่า Amplification Master Mix
47.5 µL น้ำเปล่าฟรี
5.0 µL JumpStart Taq DNA Polymerase (12.5 หน่วย) สำหรับ WGA1
- หรือ -
5.0 µL WGA DNA Polymerase สำหรับ WGA2 - ผสมให้เข้ากันปั่นเหวี่ยงเป็นเวลาสั้นๆและเริ่มการปั่นด้วยความร้อน
- หลังจากปั่นจักรยานเสร็จแล้วให้รักษาปฏิกิริยาที่อุณหภูมิ 4°C หรือเก็บที่อุณหภูมิ– 20°C จนกว่าจะพร้อมสำหรับการวิเคราะห์หรือการทำให้บริสุทธิ์
การขยายเสียง
- เติมดีเอ็นเอขยาย µL วน 1 นาโนกรัม/มล. WGA 10 มล. ลงในหลอด PCR หรือแผ่นมัลติเวล
จำเป็นต้องทำความสะอาดปฏิกิริยา WGA เพื่อลดอคติที่เป็นไปได้ในการขยายซ้ำ เราขอแนะนำให้ใช้ชุดทำความสะอาด GenElute ™ PCR (หมายเลขผลิตภัณฑ์ NA1020) หรือวิธีการทำให้บริสุทธิ์มาตรฐานที่แยกดีเอ็นเอเดี่ยวและคู่ที่ติดอยู่ - สร้างการผสมผสานการขยายเสียง สำหรับปฏิกิริยาการขยายแต่ละครั้งให้เพิ่มสิ่งต่อไปนี้ลงใน DNA ที่ขยายของ WGA (ขั้นตอนที่ 1):
47.5 µL ของน้ำที่ไม่มี
การปล่อยนิวเคลียร์ 7.5 µL ของ 10 X Amplification Master Mix
5 µL ของ WGA DNA Polymerase - Vortex อย่างละเอียด, ปั่นเหวี่ยงสั้นๆและเริ่มต้น thermycycling โปรไฟล์ต่อไปนี้ได้รับการปรับให้เหมาะสมสำหรับ PE 9700 หรือเทอร์โมไซเซอร์ที่เทียบเท่า:
การลดความอิ่มตัวเริ่มต้น 95°C เป็นเวลา 3 นาที
ทำ 14 รอบดังนี้:
ความสูง 94°C เป็นเวลา 15 วินาที
หลอม/ขยาย 65°C เป็นเวลา 5 นาที - หลังจากปั่นจักรยานเสร็จแล้วให้รักษาปฏิกิริยาที่อุณหภูมิ 4°C หรือเก็บที่อุณหภูมิ– 20°C จนกว่าจะพร้อมสำหรับการวิเคราะห์หรือการทำให้บริสุทธิ์ ความเสถียรของ WGA DNA เทียบเท่ากับ DNA จีโนมิกที่เก็บไว้ภายใต้เงื่อนไขเดียวกัน
การทำให้บริสุทธิ์ของตัวอย่าง Buccal Swab ที่ขยาย
การทำให้บริสุทธิ์ของผลิตภัณฑ์ที่ขยายด้วย GenElute PCR ชุดทำความสะอาด(NA1020)
- ใส่คอลัมน์เย็บเล่ม GenElute Miniprep (ที่มีโอริงสีน้ำเงิน) ลงในหลอดเก็บตัวอย่างที่ให้มาหากยังไม่ได้ประกอบ เติมสารละลายเตรียมคอลัมน์ 0.5 มล. ลงในคอลัมน์ miniprep แต่ละคอลัมน์และปั่นเหวี่ยงที่แรงเหวี่ยง 12,000 x g เป็นเวลา 30 วินาทีถึง 1 นาที ทิ้ง Eluate
โซลูชันการเตรียมคอลัมน์ช่วยเพิ่มการยึดเกาะดีเอ็นเอให้กับเมมเบรนทำให้ได้ผลผลิตที่สม่ำเสมอมากขึ้น - เพิ่มสารละลายที่มีผลผูกพัน 5 ปริมาตรลงในปริมาตร 1 ของปฏิกิริยา PCR และการผสม ตัวอย่างเช่นเพิ่มสารละลายที่มีผลผูกพัน 500 µL ลงใน 100 µL ของปฏิกิริยา PCR ถ่ายโอนโซลูชันลงในคอลัมน์การเชื่อมโยง ปั่นเหวี่ยงคอลัมน์ด้วยความเร็วสูงสุด (12,000 ถึง 16,000 x g) เป็นเวลา 1 นาที ทิ้งหลอดดูดเลือดแต่เก็บหลอดเก็บตัวอย่างไว้
- ใส่แกนที่เย็บเข้าไปในหลอดเก็บตัวอย่าง ใช้น้ำยาทำความสะอาดเจือจาง 0.5 มล. กับคอลัมน์และปั่นเหวี่ยงด้วยความเร็วสูงสุด 1 นาที ทิ้งหลอดดูดเลือดแต่เก็บหลอดเก็บตัวอย่างไว้
ตรวจสอบให้แน่ใจว่าได้เติมเอทานอลลงในน้ำยาล้างน้ำยาเข้มข้นก่อนใช้ครั้งแรก โปรดดูคำแนะนำในการเตรียมการ - ใส่คอลัมน์กลับเข้าไปในหลอดเก็บตัวอย่าง ปั่นเหวี่ยงคอลัมน์ที่ความเร็วสูงสุดเป็นเวลา 2 นาทีโดยไม่ต้องใช้น้ำยาล้างเพิ่มเติมเพื่อขจัดเอทานอลส่วนเกิน ทิ้งอิลูเอตที่เหลืออยู่รวมทั้งหลอดเก็บตัวอย่าง
- ถ่ายคอลัมน์ลงในหลอดเก็บตัวอย่างขนาด 2 มล. ที่สดใหม่ ใช้สารละลายเจือจาง 50 μ m หรือ µL ที่ตรงกลางของแต่ละคอลัมน์ บ่มเชื้อที่อุณหภูมิห้องเป็นเวลา 1 นาที
เมื่อใช้น้ำในการเดินทางตรวจสอบให้แน่ใจว่าค่า pH ของน้ำอยู่ระหว่าง 5.5 ถึง 8.5 นอกจากนี้ยังสามารถใช้สารละลายเจือจางเจือจางด้วยน้ำได้ 10 เท่า - ในการกำจัดดีเอ็นเอให้ปั่นเหวี่ยงคอลัมน์ด้วยความเร็วสูงสุด 1 นาที ขณะนี้ผลิตภัณฑ์ขยาย PCR มีอยู่ใน eluate และพร้อมสำหรับการใช้งานทันทีหรือจัดเก็บที่อุณหภูมิ– 20°C
การวัดปริมาณของตัวอย่าง Buccal Swab ที่ขยาย
ปริมาณของ DNA ที่ขยายโดยใช้การขยายจีโนมทั้งหมดสามารถตรวจจับได้โดยมีหรือไม่มีการทำให้บริสุทธิ์ สำหรับตัวอย่างที่มีคุณภาพสูงสุดของดีเอ็นเอขอแนะนำอย่างยิ่งให้ทำความสะอาดตัวอย่างหลังจากการขยาย ผลิตภัณฑ์ที่ขยายสามารถวัดได้ด้วย PicoGreen® dsDNA Quantitation Assay จาก Molecular Probes Inc. (7589) อีกวิธีหนึ่งในการตรวจจับผลิตภัณฑ์ที่มีการขยายคือการดูดกลืนสเปกโตรโฟโต เมตริก (OD260) บนสเปกโตรโฟโตมิเตอร์ NanoDrop ® เครื่องมือนี้สามารถ μL การวัดการดูดซับของตัวอย่าง 1 μ m ในช่วงไดนามิกขนาดใหญ่ 2 (37-3,700 นาโนกรัม/μL)
ข้อมูลแอปพลิเคชันสำหรับการขยายตัวอย่าง Buccal Swab
การขยายจีโนมทั้งหมดดำเนินการกับตัวอย่างสวาบของ buccal
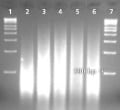
ผลิตภัณฑ์ที่ขยายจะถูกแสดงผลบนเจล agarose 1.5% 5 µL ของผลิตภัณฑ์ที่ขยายได้รับการโหลดต่อหลุม ผลิตภัณฑ์ที่ขยาย GenomePlex ทำให้มีขนาดเฉลี่ย 400 bp รูปแบบรอยเปื้อนจะแตกต่างกันไปตามแหล่งที่มาตามที่แสดงบนเจล
เลน 1 - 1 kb บันได
เลน 2 - Blood
Lane 3 - Plant
Lane 4 - Buccal Swab
Lane 5 - Soil
Lane 6 - Positive Control Lane
7 - 1 kb Ladder
วัสดุ
ต้องมีเอกสารเพิ่มเติม
- สแวบตัวเล็ก
- หลอดเก็บตัวอย่างขนาดเล็ก 1.5 มล
- เครื่องปั่นแยกขนาดเล็ก (พร้อมโรเตอร์สำหรับหลอดขนาด 2 มล.)
- อ่างน้ำหรือบล็อกความร้อน 55°C
เพื่ออ่านต่อ โปรดเข้าสู่ระบบหรือสร้างบัญชีใหม่
ยังไม่มีบัญชีใช่หรือไม่?