การแก้ไขปัญหา RT-PCR
ในหน้านี้
- การพัฒนาโปรโตคอลการแก้ไขปัญหา
- การปรับแต่งการออกแบบ Oligo
- การเพิ่มประสิทธิภาพการวิเคราะห์ PCR
- การออกแบบการวิเคราะห์ RT-PCR และ QPCR
- คุณภาพเทมเพลต RNA หรือ DNA
- โปรแกรม PCR การวิเคราะห์
- Thermo Cycler ทำงานผิดปกติ
- Troubleshooting Examples: Diagnostic Tools
- การแยกตัว/ละลายเส้นโค้ง
- การแก้ไขปัญหากรณีศึกษา RT-PCR
- สรุป—รายการตรวจสอบการแก้ไขปัญหา PCR
การพัฒนา PCR หรือ RT-PCR Troubleshooting Protocol
สาเหตุที่เป็นไปได้ของข้อผิดพลาดและ/หรือปัญหาข้อผิดพลาดของผู้ปฏิบัติงาน
มีความเป็นไปได้มากมายสำหรับข้อผิดพลาดของผู้ควบคุม แหล่งที่มาของข้อผิดพลาดเหล่านี้มักไม่สามารถระบุได้ ขั้นตอนแรกในขั้นตอนการแก้ไขปัญหาใดๆคือการตรวจสอบโปรโตคอลและทำการทดสอบซ้ำ การตรวจสอบโปรโต คอล (ดูภาคผนวก A, โปรโตคอลของคู่มือนี้) และขอให้นักชีววิทยาโมเลกุลที่มีประสบการณ์ทบทวนแผนการทดลองเป็นสิ่งสำคัญ เรื่องราวข้อควรระวังของเพื่อนหลังปริญญาเอกที่วิ่ง PCR ที่ล้มเหลวหลายครั้งก่อนที่จะตระหนักว่า dNTPs หายไปจาก ต้นแบบ PCR Mi xis เตือนว่าแม้แต่นักวิทยาศาสตร์ที่ทำงานมากเกินไปที่ดีที่สุดก็มีความเสี่ยงต่อข้อผิดพลาดง่ายๆ
มาสเตอร์มิกซ์
ความผิดพลาดหรือปัญหากับส่วนผสมหลักของส่วนประกอบปฏิกิริยาอาจเป็นแหล่งที่มาของความล้มเหลวร้ายแรงของการขยายในตัวอย่างทั้งหมดและการควบคุมเชิงบวก ก่อนที่จะทำการทดสอบซ้ำให้ตรวจสอบส่วนประกอบทั้งหมดและความเข้มข้นของส่วนประกอบเหล่านั้น หากมีการใช้น้ำยาชุดใหม่จะเป็นการป้องกันที่มีประโยชน์ในการรันน้ำยาชุดใหม่ก่อนที่จะเริ่มใช้งานในชุดการทดลองที่สำคัญ
เมื่อเปลี่ยนผลิตภัณฑ์หลักผสมเป็นสิ่งสำคัญที่จะต้องตระหนักว่าการวิเคราะห์บางอย่างมีความไวเป็นพิเศษต่อการจัดองค์ประกอบบัฟเฟอร์/อุณหภูมิการหลอม(Ta)/การผสมความเข้มข้นของไพรเมอร์ การเปลี่ยนแปลงสิ่งใดสิ่งหนึ่งเหล่านี้อาจส่งผลให้เกิดประสิทธิภาพที่แตกต่างกัน ดังนั้นให้ตรวจสอบการวิเคราะห์ทั้งหมดในการผสมมาสเตอร์ที่เลือกและบนเครื่องมือที่ต้องการทั้งหมดก่อนที่จะทำการเปลี่ยนแปลงที่รุนแรง นอกจากนี้ยังจำเป็นต้องทบทวนคำแนะนำที่มาพร้อมกับส่วนผสมหลักแต่ละชนิดเนื่องจากระบุเงื่อนไขที่แนะนำซึ่งเหมาะสำหรับเอนไซม์กลไกการสตาร์ทร้อนและส่วนประกอบบัฟเฟอร์ที่กำหนด
เป็นการปฏิบัติที่ดีในห้องปฏิบัติการเพื่อให้แน่ใจว่ามีการเตรียมส่วนผสมหลักของปฏิกิริยาเพียงพอสำหรับตัวอย่างทั้งหมดที่จะใช้ร่วมกัน ตรวจสอบให้แน่ใจว่าส่วนประกอบทั้งหมดได้รับการละลายและผสมอย่างดีและส่วนผสมหลักของการทดลองผสมกันอย่างดีก่อนที่จะแบ่งตัวอย่าง ซึ่งเกี่ยวข้องกับบัฟเฟอร์ 2 × บางส่วนเช่น KiCqStart® ที่มีความหนืดมากกว่าบัฟเฟอร์ PCR ปกติ
Oligo Optimization
Oligos อาจทำให้เกิดปัญหาหากเป็นลำดับที่ไม่ถูกต้องหรือออกแบบมาไม่ดีทำงานที่ความเข้มข้นต่ำกว่าที่เหมาะสม Ta ย่อยที่เหมาะสมหรือติดฉลากไม่เพียงพอหรือดับ (สำหรับโพรบ) การทดสอบที่ทำงานภายใต้สภาวะที่เหมาะสมที่สุดสำหรับ oligo หรือการใช้การออกแบบที่ไม่ดีอาจทำให้ได้ข้อมูลบางอย่างแต่อาจไม่ได้สะท้อนถึงชีววิทยาของแท้ภายใต้การพิจารณา เมื่อได้รับ oligo lyophilized มันเป็นสิ่งสำคัญที่จะ:
- ตรวจสอบลำดับ
- Ensure that all DNA is resuspended prior to use
- ยืนยันว่าสารละลายมีความเข้มข้นตามที่คาดไว้
resuspend oligos โดยการให้ความร้อน oligo ถึง 90°C เป็นเวลา 5 นาทีแล้วผสมให้เข้ากัน รอบการละลายน้ำแข็งแบบแช่แข็งซ้ำๆอาจส่งผลต่อประสิทธิภาพการทำงานของ oligo ดังนั้น oligos ทั้งหมดที่ความเข้มข้นของสต็อก (โดยปกติ 100 μM) ควรจะเป็น aliquoted และเก็บไว้ที่ -20°C หรือ -80°C ในระยะยาว
ในระหว่างขั้นตอนการแก้ไขปัญหาจำเป็นอย่างยิ่งที่จะต้องตรวจสอบว่าลำดับที่ถูกต้องได้รับการสั่งซื้อโดยกลับไปที่ลำดับเป้าหมายและยืนยันว่ามีลำดับ oligo อยู่จริง ตรวจสอบให้แน่ใจว่าคุณภาพของ oligo ถูกต้องโดยติดต่อผู้ขาย oligo วัดความเข้มข้นในการทำงานของ oligo และตรวจสอบโมเลกุลฟลูออเรสเซนต์ด้วยสายตาเพื่อยืนยันว่ามีการติดฉลาก ทดสอบไพรเมอร์ของการวิเคราะห์โพรบใน ส่วนผสม SYBR ® Green I qPCR เพื่อตรวจสอบการขยาย พิจารณาการปรับความเข้มข้นของไพรเมอร์หรือ Ta ให้เหมาะสม (ดูที่ การปรับประสิทธิภาพและการตรวจสอบการวิเคราะห์) เมื่อใช้โพรบเป็นครั้งแรกให้รวบรวมข้อมูลฟลูออเรสเซนต์สำหรับความยาวคลื่นที่อาจเกิดขึ้นให้มากที่สุดเท่าที่จะเป็นไปได้เพื่อให้สามารถตรวจสอบการรั่วไหลของสัญญาณระหว่างช่องและสามารถตรวจจับความผิดพลาดในการติดฉลากได้
การเพิ่มประสิทธิภาพ PCR ไม่เพียงพอ
ผลของการเพิ่มประสิทธิภาพการทดสอบได้รับการอธิบายและแสดงให้เห็นใน การเพิ่มประสิทธิภาพการทดสอบและการตรวจสอบ เมื่อการวิเคราะห์ล้มเหลวหรือทำงานได้ดีกว่าแต่ไม่มีข้อผิดพลาดในการออกแบบหรือขั้นตอนการดำเนินงานอาจได้รับประโยชน์จากการเพิ่มประสิทธิภาพของเงื่อนไขการทดลอง เมื่อทำการแก้ไขปัญหาให้ทดสอบไพรเมอร์ที่ความเข้มข้นขั้นสุดท้าย 100 Nm, 500 Nm และ 900 Nm และ/หรือ Ta ระหว่าง 55°C ถึง 70°C (โดยใช้การไล่ระดับอุณหภูมิ) เพื่อระบุว่าการวิเคราะห์จะปรับปรุงด้วยการเพิ่มประสิทธิภาพเพิ่มเติมหรือไม่
การออกแบบการวิเคราะห์ RT-PCR และ QPCR
การออกแบบการวิเคราะห์ได้รับการอธิบายไว้ใน การออกแบบการวิเคราะห์ PCR/qPCR/dPCR เมื่อแก้ไขปัญหาการวิเคราะห์ตรวจสอบให้แน่ใจว่าการออกแบบได้รับการตรวจสอบแล้ว ยืนยันว่าตำแหน่งไพรเมอร์ PCR/qPCR และแอมพลิฟายเออร์สอดคล้องกับโปรโตคอลการล่อน้ำมัน RT ตัวอย่างเช่นตรวจสอบให้แน่ใจว่าการวิเคราะห์ที่ใช้กับ cDNA ที่เตรียมไว้หลังจากการเตรียมรองพื้น oligo-DT นั้นอยู่ที่ 3’ของทรานสคริปท์ ตรวจสอบให้แน่ใจว่าข้อมูลลำดับมีความน่าเชื่อถือและมีการพิจารณาสายพันธุ์การต่อที่เหมาะสมและ SNPs
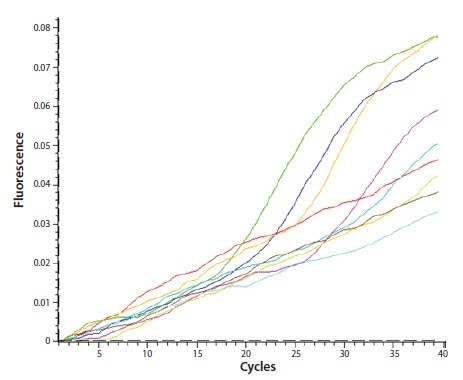
รูปที่ 11 1 กการวิเคราะห์มีรายละเอียดพล็อตการขยายที่ผิดปกติโดยมีการเบี่ยงเบนที่เด่นชัดของเบสไลน์

รูปที่ 11 1 บาทลำดับของโพรบที่รวมอยู่ในการวิเคราะห์ถูกป้อนลงในซอฟต์แวร์คาดการณ์การพับแบบพับ เป็นที่ชัดเจนว่าโพรบสามารถใช้โครงสร้างพับที่มั่นคงในการแก้ปัญหาและอาจส่งผลให้เกิดปัญหาที่สังเกตได้
คุณภาพเทมเพลต RNA หรือ DNA
ผลกระทบของคุณภาพเทมเพลตในประสิทธิภาพการทดสอบได้รับการอธิบายไว้ใน การทำให้บริสุทธิ์ตัวอย่างและการประเมินคุณภาพ คุณภาพเทมเพลตประกอบด้วยการพิจารณาปริมาณความสมบูรณ์และการมีสารยับยั้ง จำเป็นอย่างยิ่งที่จะต้องตรวจสอบให้แน่ใจว่าคุณภาพ RNA ตรงกับโปรโตคอล การเตรียม RTที่เหมาะสมที่สุด (ดูการถอดสคริปต์ย้อนกลับ) และใช้เทมเพลตคุณภาพดีที่สุดเท่าที่จะเป็นไปได้ ในทำนองเดียวกันปริมาณของ RNA ที่เพิ่มลงในปฏิกิริยา RT จะต้องอยู่ภายในขอบเขตของโปรโตคอลและในหลายกรณีนี่ควรเหมือนกันสำหรับปฏิกิริยาทั้งหมด ReadyScript® เป็นข้อยกเว้นที่โดดเด่นสำหรับแนวทางนี้เนื่องจากการนำน้ำยาและโปรโตคอลนี้มาใช้จะให้ความเข้มข้น cDNA เชิงเส้นที่เป็นสัดส่วนกับปริมาณ RNA อินพุต เมื่อทำการแก้ไขปัญหาตัวอย่างที่ให้ปริมาณ Cq สูงกว่าที่คาดไว้ให้ ทำการทดสอบ SPUD หรือเจือจางตัวอย่างผ่านชุดการเจือจาง 1: 11 หรือ 1: 2 และทำการทดสอบซ้ำ (รูปที่ 1.1) เพื่อระบุตัวอย่างที่มีสารยับยั้ง


รูปที่ 11 2การขยายตัวของการเจือจางแบบอนุกรม 10 เท่าของแม่แบบ DNA การทำซ้ำมีความแม่นยำแต่ n ü ΔCq ไม่สอดคล้องกันลดลงด้วยการเจือจางที่เพิ่มขึ้น ข้อมูลนี้ยังแสดงสัญญาณบวกในการควบคุมเทมเพลต NO (NTC) ที่ระบุการปนเปื้อนหรือการสร้างตัวปรับสีรองพื้นและการเจือจางน้อยกว่า 105 สำเนามีข้อมูลที่เหมือนกันกับ NTC
ปริมาณเทมเพลตยังเป็นสิ่งสำคัญที่ต้องพิจารณาอีกด้วย การใส่แม่แบบลงใน PCR มากเกินไปหรือน้อยเกินไปจะส่งผลให้เกิดปฏิกิริยาที่ล้มเหลวและแผนการขยาย qPCR ที่ผิดปกติ รูปที่ 11.3 แสดงปฏิกิริยาที่มีการเจือจางแบบอนุกรม 10 เท่าของแม่แบบ oligo เทียม การเจือจางที่ต่ำกว่ามีความเข้มข้นมากเกินไปสำหรับปฏิกิริยาที่จะมีประสิทธิภาพหรือสำหรับเครื่องมือในการประมวลผลข้อมูลพื้นฐานได้อย่างมีประสิทธิภาพ (รูปที่ 11 3 ข) ส่งผลให้เกิดการขยายที่ผิดปกติและข้อมูลที่ไม่น่าเชื่อถือ

รูปที่ 11 3 A)การขยายการเจือจางแบบอนุกรม 10 เท่าของเทมเพลตเทียมที่มีไพรเมอร์เฉพาะและโพรบที่ติดฉลาก FAM Cq ต่ำมากสำหรับตัวอย่างที่มีความเข้มข้นแปลงการขยายไม่ได้เว้นระยะเป็นประจำและผิดปกติ b) แสดงข้อมูลดิบสำหรับแปลงขยายเหล่านี้ ปฏิกิริยาที่มีความเข้มข้นสูงสุดของชิ้นงานจะมีการเรืองแสงพื้นหลังที่สูงขึ้นอย่างมากและมีการเรืองแสงน้อยที่สุดผ่านปฏิกิริยา
โปรแกรม PCR การวิเคราะห์
เงื่อนไขการขี่จักรยาน PCR ต้องเหมาะสำหรับทั้งการทดลองและตัวทำปฏิกิริยา (เช่น ดู Master Mix) ไม่แนะนำให้ยอมรับการตั้งค่าอุปกรณ์เริ่มต้นโดยไม่มีการตรวจสอบ
ความผิดปกติของไซโคลนความร้อน
ความผิดพลาดของเครื่องมืออาจมีการโจมตีอย่างรุนแรงและอาจทำให้วินิจฉัยได้ยาก เพื่อป้องกันค่าใช้จ่ายในการซ่อมแซมที่มีราคาแพงตรวจสอบให้แน่ใจว่าผู้ปฏิบัติงานทุกรายได้รับการฝึกอบรมและดูแลอย่างเต็มที่ ความผิดพลาดของเครื่องมือบางอย่างทำให้เกิดความล้มเหลวร้ายแรงส่งผลให้ไม่มีการขยายหรือข้อมูลเรืองแสงในขณะที่บางอย่างบิดเบือนข้อมูลหรือรักษาตัวอย่างในลักษณะที่ไม่สม่ำเสมอสร้างความแตกต่างเทียมระหว่างตัวอย่างทางชีวภาพที่เหมือนกัน การใช้ตัวอย่างควบคุมที่มีการวิเคราะห์การควบคุมเป็นสิ่งที่ประเมินค่าไม่ได้สำหรับการแก้ไขปัญหา เมื่อสงสัยว่าอุปกรณ์มีความผิดปกติควรทำการทดสอบที่เชื่อถือได้และเหมาะสมที่สุดในทุกหลุม การตรวจสอบความสม่ำเสมอนี้จะเปิดเผยปัญหาที่เฉพาะเจาะจงกับภูมิภาคของเครื่องมือรวมถึงปัญหาการวิเคราะห์และเครื่องมือแยกต่างหาก
ตัวอย่างการแก้ไขปัญหา PCR แสดงการใช้เครื่องมือการวินิจฉัย
หลังจากรัน PCR ที่วางแผนไว้เป็นอย่างดีแล้วจะมีเครื่องมือการวินิจฉัยหลายอย่างที่พร้อมใช้งานสำหรับการแก้ไขปัญหา:
- ควบคุมตัวอย่างและการวิเคราะห์
- เจลจุดสิ้นสุด/SYBR สีเขียว I ย้อมรีเอเจนต์
- แพล๊อตขยาย (ตรวจสอบโปรไฟล์พล็อตซ้ำและการขยาย)
- เส้นโค้งมาตรฐาน (เกรเดียนท์และ R2)/ ชุดการเจือจาง
- จุดหลอมเหลว/การแยกตัว (SYBR Green I Dye, Molecular Beacons, Scorpions® Probes)
- มุมมองข้อมูลดิบ/หลายองค์ประกอบ
ควบคุมตัวอย่าง/ปฏิกิริยา
ขอแนะนำให้ใช้ตัวควบคุมเป็นอย่างยิ่ง แทบจะเป็นไปไม่ได้เลยที่จะแก้ไขปัญหาการวิเคราะห์ที่ล้มเหลวโดยไม่มีข้อมูลจากชุดการควบคุมที่เหมาะสม
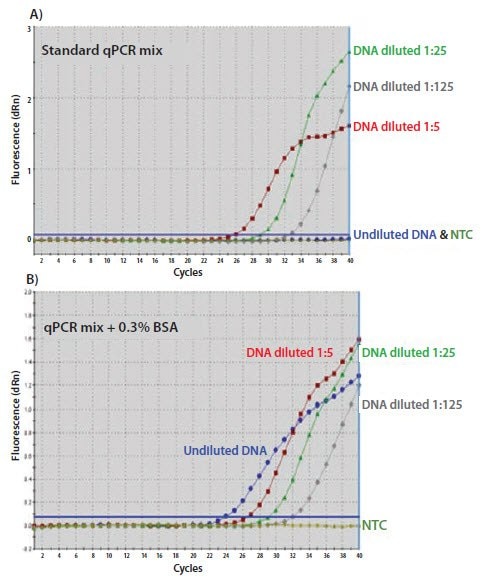
รูปที่ 11 4 A)เทมเพลตที่ไม่เจือจางไม่สามารถขยายได้ในขณะที่การเจือจางแสดงประสิทธิภาพการขยายที่ดีขึ้น b) การเพิ่ม BSA 0.3% ลงในการผสม qPCR จะสนับสนุนการขยายจากเทมเพลตที่ไม่มีการเจือจาง
การตรวจสอบการวิเคราะห์ที่ล้มเหลวอย่างสมบูรณ์อาจเป็นเรื่องยากเนื่องจากมีข้อมูลเพียงเล็กน้อยในการแก้ไขปัญหา เนื่องจากความล้มเหลวในการวิเคราะห์หลายอย่างเป็นผลมาจากข้อผิดพลาดร้ายแรงบางอย่างการตรวจสอบครั้งแรกควรเป็นการตรวจสอบการตั้งค่าการทดสอบแล้วทำซ้ำ PCR หากล้มเหลวกระบวนการแก้ไขปัญหาจะขึ้นอยู่กับข้อมูลเกี่ยวกับแต่ละส่วนประกอบของการทดสอบ (รูปที่ 11-5)

รูปที่ 11 5ขั้นตอนการแก้ไขปัญหาเบื้องต้นสำหรับ PCR
เมื่อการทดสอบ qPCR ล้มเหลวอย่างสมบูรณ์ขั้นตอนแรกคือการตรวจสอบการออกแบบการวิเคราะห์ลำดับ oligo และข้อมูล QC จากผู้ผลิต oligo แม้ว่าการวิเคราะห์อาจล้มเหลวแต่สามารถใช้ข้อมูล QPCR แบบหลายส่วนประกอบ/ข้อมูลดิบเพื่อให้ข้อมูลเพิ่มเติมได้ รูปที่ 11.6 แสดงพลอตข้อมูลดิบสำหรับการวิเคราะห์สองครั้งที่มีโพรบที่ติดฉลากว่า 6 FAM ™หรือ HEX ™ (VIC ®) แม้ว่าการวิเคราะห์ทั้งสองจะแสดงการขยายสัญญาณแต่สัญญาณฐานสิบหกจะอยู่ที่ประมาณครึ่งหนึ่งของสัญญาณ FAM เนื่องจากนี่เป็นสีย้อมที่อ่อนแอกว่าโดยเนื้อแท้นี่จึงเป็นการสังเกตปกติ การวิเคราะห์เจล agarose (รูปที่ 11 6 ข) แสดงให้เห็นว่าปฏิกิริยาทั้งสองมีความเข้มข้นใกล้เคียงกันของผลิตภัณฑ์สนับสนุนการสังเกตว่า ค่า qPCR cq มีความคล้ายคลึงกัน

รูปที่ 11 6 A)พลอตข้อมูลดิบของการวิเคราะห์แบบดูเพล็กซ์ที่มี FAM และโพรบ HEXlabeled โพรบ FAM ให้การเรืองแสงที่สูงขึ้นอย่างเป็นธรรมชาติ b) เจล agarose ที่แสดงว่ามีการผลิตผลิตภัณฑ์ในปริมาณที่เท่ากันในแต่ละปฏิกิริยาและยืนยันการสังเกต qPCR cq
การตรวจสอบข้อมูลดิบเป็นการตรวจสอบที่มีประโยชน์ในการตรวจสอบว่าโพรบได้รับการติดฉลากอย่างถูกต้องและถูกเพิ่มลงในปฏิกิริยา รูปที่ 11 7 แสดงข้อมูลดิบสำหรับการขยายเป้าหมายสามเป้าหมายในการทดลองสามเท่า โพรบเฉพาะสำหรับแต่ละชิ้นงานจะมีป้ายกำกับ FAM, HEX และ TAMRA โพรบ HEX และ TAMRA แสดงพื้นหลังที่ต่ำและการขยายที่มีประสิทธิภาพแต่สัญญาณ FAM สูงอย่างต่อเนื่องตลอดการทดลองและไม่มีหลักฐานของการขยาย ซึ่งสอดคล้องกับความเข้มข้นของโพรบที่สูงเกินไปในปฏิกิริยาหรือความผิดพลาดกับโพรบโดยไม่มีการดับสัญญาณในครั้งแรก ในกรณีดังกล่าวควรตรวจสอบความเข้มข้นของโพรบและการออกแบบการวิเคราะห์เพื่อให้แน่ใจว่าโพรบมีฉลากและดับที่เข้ากันได้และหากจำเป็นให้ทดสอบโพรบใหม่

รูปที่ 11 7มีการใช้ปฏิกิริยาสามเท่าในการตรวจจับชิ้นงานสามชิ้นโดยใช้โพรบที่มีฉลาก FAM, HEX และ TAMRA โพรบ HEX และ TAMRA ให้การขยายจากเป้าหมายแต่โพรบ FAM ไม่มีการขยาย การตรวจสอบข้อมูลดิบแสดงให้เห็นว่าการเรืองแสงพื้นหลังสูงเป็นพิเศษและไม่มีการสังเกตความแตกต่างตลอดการเกิดปฏิกิริยา ซึ่งสอดคล้องกับความเข้มข้นของโพรบที่สูงเกินไปในปฏิกิริยาหรือโพรบที่มีข้อบกพร่องที่มีการดับไม่เพียงพอ
หากการทดสอบเดิมที่ใช้ในการตรวจจับโพรบควรทำการทดสอบซ้ำโดยใช้ตัวทำปฏิกิริยา SYBR Green I รวมถึงการควบคุมเชิงบวกและลบ (แต่ไม่ใช่ตัวอย่างที่มีค่า) หรือสามารถตรวจสอบผลิตภัณฑ์ของปฏิกิริยาที่ล้มเหลวได้บนเจล agarose ที่ย้อมสี ethidium bromide การนำแนวทาง SYBR Green I มาใช้ในการทำการทดสอบซ้ำเป็นวิธีที่ดีกว่าเพราะจะช่วยหลีกเลี่ยงความเสี่ยงของการปนเปื้อนและให้การทดสอบซ้ำเพื่อตรวจสอบความล้มเหลวเริ่มต้น หากการทดสอบ SYBR Green I ให้ข้อมูลอาจเป็นไปได้ว่าความล้มเหลวของโพรบเดิมเกิดจากข้อผิดพลาดทางเทคนิคหรือความผิดพลาดของโพรบ ในการแยกความแตกต่างระหว่างข้อผิดพลาดในการทดลองหรือความผิดพลาดกับโพรบให้ทำการทดสอบโพรบซ้ำอีกครั้งหากปฏิกิริยาล้มเหลวอีกครั้งให้เปลี่ยนโพรบ วิธีการนี้สามารถนำมาใช้เพื่อตรวจสอบปฏิกิริยาที่ทำให้เกิดข้อมูลที่ไม่ดีได้ ในตัวอย่างที่แสดงใน รูปที่ 11.8ปฏิกิริยาของโพรบนั้นน้อยที่สุดและเมื่อเทียบกับปฏิกิริยาที่รันโดยใช้ SYBR Green I จะเห็นได้ว่าสัญญาณโพรบไม่สะท้อนการทดลอง ในกรณีดังกล่าวการออกแบบการวิเคราะห์ควรได้รับการตรวจสอบและการทดสอบโพรบใหม่

รูปที่ 11 8ปฏิกิริยาที่เหมือนกันถูกเรียกใช้โดยมีโพรบ qPCR หรือสีย้อม SYBR Green I (ตามที่ระบุ) ปฏิกิริยา SYBR Green I มีความไวมากขึ้นประมาณสิบเอ็ดรอบและให้ฟลูออเรสเซนซ์จุดปลายที่สูงขึ้นมาก นี่เป็นการบ่งชี้ถึงความผิดพลาดในการสอบสวนหรือปัญหาเกี่ยวกับการออกแบบโพรบ (ข้อมูลที่ได้รับจากศาสตราจารย์ Stephen Bustin, UK)
กำลังตรวจสอบการติดฉลากโพรบ
ข้อมูลดิบหรือพล็อตหลายองค์ประกอบเป็นเครื่องมือวินิจฉัยที่มีประโยชน์ในการตรวจสอบว่ามีความเข้มข้นของโพรบที่เหมาะสมรวมอยู่ในปฏิกิริยาหรือไม่และโพรบได้รับการติดฉลากและดับอย่างเพียงพอหรือไม่ รูปที่ 11 9 แสดงพล็อตหลายองค์ประกอบสำหรับปฏิกิริยาที่มีสามโพรบ สองครั้งแรกสร้างแปลงขยายและเรืองแสงพื้นหลังจะเห็นได้ชัด ไม่มีข้อมูลจากโพรบตัวที่สามและการตรวจสอบข้อมูลดิบแสดงให้เห็นว่าการเรืองแสงพื้นหลังเทียบเท่ากับการควบคุมน้ำเปล่าซึ่งไม่มีโพรบ ดังนั้นข้อมูลนี้จึงเป็นผลมาจากการไม่มีฟลูออเรสเซนซ์ในปฏิกิริยา ซึ่งอาจเกิดจากข้อผิดพลาดในระหว่างการตั้งค่าที่ไม่ได้ให้โพรบมาหรือโพรบไม่ได้ติดฉลากไว้

รูปที่ 11 9ตรวจพบยีนสามตัวในตัวอย่างเทมเพลตเดียวกัน ปฏิกิริยาสองครั้งส่งผลให้เกิดการขยายสัญญาณ (1 และ 2 อย่างไรก็ตามครั้งที่ 3 เป็นลบ การตรวจสอบมุมมองหลายองค์ประกอบจะแสดงให้เห็นว่าการเรืองแสงพื้นหลังสำหรับปฏิกิริยาที่ 3 นั้นเทียบเท่ากับการควบคุมน้ำที่ระบุว่าไม่มีสัญญาณ
การตรวจสอบการติดฉลากโพรบเพิ่มเติมสามารถทำได้โดยใช้การย่อย DNase I ซึ่งจะต้องดำเนินการด้วยความระมัดระวังอย่างมากเพื่อให้แน่ใจว่าโพรบและหุ้นไพรเมอร์จะไม่ปนเปื้อนกับเอนไซม์ซึ่งจะนำไปสู่ผลลัพธ์ที่ร้ายแรง อะลิควอทของโพรบที่ล้มเหลว (รูปที่ 11A) เทียบเท่ากับที่รวมอยู่ในปฏิกิริยาเช่น 10 300 นิวตันเมตรจะถูกบ่มด้วยและไม่มี DNase I ซึ่งสามารถดำเนินการได้แบบเรียลไทม์ (รูปที่ 11 10 บี) เพื่อให้วัดปริมาณฟลูออเรสเซนต์ได้ตามเวลาหรืออีกทางเลือกหนึ่งคือจุดเริ่มต้นและจุดสิ้นสุด (หลังจาก 10 นาที) จะให้ข้อมูลที่เพียงพอ เมื่อทำการทดสอบนี้สิ่งสำคัญคือต้องเปรียบเทียบข้อมูลกับโพรบที่ทำงานได้ดีและมีฉลากเรืองแสงและดับเพลิงเดียวกัน (รูปที่ 11 10 บี)

รูปที่ 11 10 A)ตรวจพบสองเทมเพลตโดยใช้โพรบที่แตกต่างกันทั้ง FAM ที่ติดฉลากไว้ ในขณะที่การตรวจจับโดยใช้โพรบเพียงตัวเดียวส่งผลให้เกิดสัญญาณฟลูออเรสเซนต์สูงส่วนที่สองจะอ่อนแอลงมาก ข) ตัวควบคุมและโพรบทดสอบ 300 นิวตันเมตร) ถูกบ่มที่อุณหภูมิ 37°C ในเครื่องมือแบบเรียลไทม์ในบัฟเฟอร์ DNase I ในการมีหรือไม่มีเอนไซม์ DNase I การปล่อยฟลูออเรสเซนต์จากโพรบ 1 ประมาณสองเท่าจากโพรบ 2 แสดงให้เห็นว่าการติดฉลากโพรบ 2 ไม่เพียงพอ
แพล๊อตขยาย
โครงสร้างของแผนการขยายและความสามารถในการทำซ้ำทางเทคนิคให้ข้อมูลที่มากมายเกี่ยวกับคุณภาพของการทดสอบ qPCR และอาจให้ข้อบ่งชี้เตือนครั้งแรกว่าทั้งหมดไม่ได้เป็นอย่างที่ควรจะเป็น แปลงขยายใน รูปที่ 11.11 Aare ไม่ปกติมีเสียงดังมากและยากที่จะตีความอย่างถูกต้อง การตรวจสอบเพิ่มเติมของค่าการเรืองแสง DR แสดงให้เห็นว่าอัตราผลตอบแทนของฟลูออเรสเซนต์ที่จุดปลายมีเพียง 400 หน่วยซึ่งบ่งชี้ว่าปฏิกิริยาไม่เพียงพอแต่การขยายได้รับการสร้างขึ้นโดยซอฟต์แวร์เครื่องมือและปรับขนาดอัตโนมัติ ในทำนองเดียวกันข้อมูลใน รูปที่ 11.11 Bhave foxtail เด่นชัด (เส้นโค้งลดลง) ที่จุดเริ่มต้นของโปรไฟล์ก่อนที่จะเพิ่มขึ้นอีกครั้งหลังจากส่วนข้อมูลขั้นต้น การปรากฏตัวของหางปลามีความสอดคล้องกันในปฏิกิริยาทั้งสองแต่ปฏิกิริยาหนึ่งมีจุดปลายที่ต่ำกว่ามาก (รูปที่ 11 11 องศาเซลเซียส) ส่งผลให้หางปลาที่ขยายตัวและมีความสัมพันธ์กัน
รูปที่ 11 11 ก) แปลงขยายที่มีเสียงดังเนื่องจากการปรับขนาดอัตโนมัติโดยซอฟต์แวร์เครื่องมือของข้อมูลที่ไม่ดีที่มีการเรืองแสงต่ำ ข) ปฏิกิริยาที่ให้ผลลัพธ์จุดปลายต่ำ DR มี foxtail เริ่มต้นที่เด่นชัด c) FOXTAIL จะถูกมองว่าเป็นผลปกติเมื่อเป็นสัดส่วนกับการวิเคราะห์ที่มีคุณภาพสูง
ในทำนองเดียวกันแผนการขยายใน รูปที่ 11.12 Aare ผิดปกติอย่างชัดเจนและไม่สามารถใช้งานได้ตามที่แสดง พล็อตการขยายที่ลดลงต่ำกว่า11 DR (รูปที่ 12A) เป็นตัวบ่งชี้คลาสสิกของการตั้งค่าพื้นฐานที่ไม่เหมาะสม การตรวจสอบข้อมูลดิบสำหรับปฏิกิริยานี้ (รูปที่ 11 12 ข) แสดงให้เห็นว่าแผนการขยายจริงมีโปรไฟล์ปกติยืนยันว่าข้อมูลที่วิเคราะห์เป็นผลมาจากปัญหาซอฟต์แวร์เครื่องมือ ข้อมูลขั้นต้นที่เหมาะสมสามารถนำไปหักจากข้อมูลดิบและนำไปใช้ในซอฟต์แวร์ได้ ในกรณีนี้รอบที่ 612 ถึง 16 แสดงระยะเชิงเส้นเริ่มต้นระยะพื้นฐานของปฏิกิริยาและเมื่อนำไปใช้จะส่งผลให้เกิดการขยายตัวตามปกติ (รูปที่ 11 องศาเซลเซียส)

รูปที่ 11 12 A)แผนการขยายสัญญาณผิดปกติอย่างเห็นได้ชัดโดยมีส่วนของการจุ่มโปรไฟล์ใต้เส้นฐาน b) การตรวจสอบข้อมูลดิบแสดงให้เห็นว่าข้อมูลปฏิกิริยาเป็นไปตามที่คาดไว้ c) การตั้งค่าเบสไลน์ของเครื่องมือตามรอบที่เหมาะสมจะคืนค่าโปรไฟล์ปกติให้กับข้อมูลของพล็อตการขยายที่วิเคราะห์
โปรไฟล์พล็อตการขยายยังสามารถตีความเพื่อให้ข้อมูลเกี่ยวกับคุณภาพของการวิเคราะห์และการเพิ่มประสิทธิภาพ รูปที่ 11.1 3 แสดงความพยายามในการขยายแม่แบบการเจือจางแบบอนุกรม 10 เท่าโดยมีความเข้มข้นแต่ละค่าที่รันใน qPCR ที่ซ้ำกัน ความสามารถในการทำซ้ำระหว่างการทำซ้ำไม่ดีความแตกต่างของรอบ (Δ Cq) ระหว่างข้อมูลไม่คงที่และไม่ใช่ 3.323 รอบตามที่คาดไว้สำหรับการเจือจางแบบอนุกรม 10 เท่า การตรวจสอบแปลงขยายโดยคำนึงถึงเส้นโค้งมาตรฐานนี้แสดงให้เห็นว่าการวิเคราะห์ต่ำกว่ามาตรฐานและไม่สามารถใช้ในการวิเคราะห์ได้ เหตุผลจะต้องมีการตรวจสอบเพิ่มเติมแต่อาจเป็นผลมาจากการออกแบบการวิเคราะห์ที่ไม่ดี (ดู PCR/qPCR/dPCR Assay Design)เงื่อนไขการวิเคราะห์ย่อยที่เหมาะสม (ดู การเพิ่มประสิทธิภาพการวิเคราะห์และการตรวจสอบ) หรือการวางท่อที่ไม่ดี (การทดสอบซ้ำ)

รูปที่ 11 13ตัวอย่าง cDNA ถูกเจือจางด้วยการเจือจางแบบอนุกรม 10 เท่าและแม่แบบเฉพาะที่ตรวจพบโดยใช้ qPCR ที่ซ้ำกันสำหรับการเจือจางแต่ละครั้ง การทำซ้ำไม่ดีแสดงถึงปัญหาเกี่ยวกับการวางท่อหรือการปรับการวิเคราะห์ให้เหมาะสม

รูปที่ 11 14ในระหว่าง qPCR มาตรฐานข้อมูลจะพุ่งขึ้นอย่างกะทันหันด้วยโปรไฟล์ที่ไม่ใช่โปรไฟล์ทั่วไป

รูปที่ 11 15สำหรับการสลายตัวหรือการเรืองแสงที่ติดยาเสพติดสาเหตุที่เป็นไปได้อาจเกิดจากเส้นใยเสริมที่แข่งขันกับไพรเมอร์และ/หรือโพรบสำหรับการหลอมแม่แบบ ไม่ต้องสนใจตราบใดที่ CT ไม่ได้รับผลกระทบ
การแยกตัว/ละลายเส้นโค้ง
การวิเคราะห์เส้นโค้งสลายตัวหรือละลายจะทำงานหลังจาก qPCR และเป็นเครื่องมือวิเคราะห์ที่ใช้ร่วมกับสีย้อมติดดีเอ็นเอ (เช่น SYBR Green I) หรือโพรบที่ไม่เสื่อมสภาพเช่น Molecular Beacons หรือ Scorpions® Probes เพื่อตรวจสอบว่ามีการขยายผลิตภัณฑ์เดียว หลังจากการขยาย PCR จะมีการบ่มไอคอนแอมพลิฟายเออร์ที่เกิดขึ้นที่อุณหภูมิที่เพิ่มขึ้นและการเปลี่ยนแปลงของสัญญาณฟลูออเรสเซนต์เมื่อการเปลี่ยนดีเอ็นเอระหว่างสถานะดับเบิลควั่นและสถานะเดี่ยวควั่น เมื่อปฏิกิริยามีแอมพลิฟายเออร์เพียงตัวเดียวจะละลายอย่างสม่ำเสมอและพล็อตของ DF/DT (อัตราการเปลี่ยนแปลงของฟลูออเรสเซนซ์เทียบกับอุณหภูมิ) จะแสดงจุดสูงสุดหนึ่งจุด การตรวจสอบเส้นโค้งละลายจะมีประสิทธิภาพโดยเฉพาะอย่างยิ่งเมื่อรวมกับข้อมูลจากการควบคุม รูปที่ 11.16 แสดงโปรไฟล์โพสต์ qPCR Melt สำหรับชุดของตัวอย่างการทดสอบทดลองการควบคุมเชิงบวกและการควบคุมแบบไม่มีเทมเพลต โปรไฟล์การละลายสำหรับตัวอย่างทดสอบจะเหมือนกับการควบคุมเชิงบวกและแต่ละตัวอย่างจะแสดงจุดสูงสุดเดียวสำหรับ DF/DT โปรไฟล์ละลายสำหรับการควบคุมแม่แบบไม่มีมีโปรไฟล์ที่กว้างขึ้นและ TMที่ต่ำกว่า การสังเกตเหล่านี้สอดคล้องกับการปรากฏตัวของไพรเมอร์ไดเมอร์ที่ปรากฏในการควบคุมเชิงลบ ซึ่งได้รับการยืนยันโดยใช้เจล agarose ย้อมสี ethidium bromide (รูปที่ 11 16 ข.) ซึ่งแสดงให้เห็นว่าไพรเมอร์จะปรากฏให้เห็นเมื่อมีเทมเพลตที่ความเข้มข้นต่ำ ซึ่งจะทำให้เกิดการประมาณการของชิ้นงานมากเกินไปเมื่อตรวจจับในตัวอย่างที่มีความเข้มข้นของชิ้นงานต่ำ ดังนั้นการวิเคราะห์ควรได้รับการปรับให้เหมาะสมหรือออกแบบใหม่ ในทางกลับกัน รูปที่ 11.16 แสดงให้เห็นว่าโปรไฟล์ละลายสำหรับผลิตภัณฑ์ในตัวควบคุมเทมเพลต NO จะเหมือนกับโปรไฟล์ละลายสำหรับการควบคุมเชิงบวกและตัวอย่างการทดสอบ นี่เป็นการบ่งชี้ที่ชัดเจนถึงการปนเปื้อนของตัวควบคุมแบบไม่มีแม่แบบที่มีแม่แบบในระหว่างการตั้งค่าการทดสอบ ตัวอย่างสุดท้ายแสดงให้เห็นถึงการรับรู้การขยายของเป้าหมายจาก gDNA ที่มีอยู่ในตัวอย่าง cDNA (รูปที่ 11 16 มิติ) แอมพลิฟายเออร์ที่ได้จาก gDNA มีความยาว
มากกว่าดังนั้นจึงมี TM ที่สูงกว่าจาก cDNA

รูปที่ 11 16 กการควบคุมเชิงบวกปฏิกิริยาการทดสอบและ NTC ถูกขยายออกไปแล้วจึงได้รับการวิเคราะห์การละลายหลังการใช้ PCR มีผลิตภัณฑ์ที่เห็นได้ชัดใน NTC ที่ละลายในอุณหภูมิที่ต่ำกว่าและมีจุดสูงสุดที่ละลายกว้างขึ้นสอดคล้องกับการก่อตัวของไพรเมอร์ไดเมอร์

รูปที่ 11 16 บาทไพรเมอร์ไดเมอร์จะเห็นได้ชัดบนความละเอียดเจลของตัวอย่างเหล่านี้ (พร้อมกับตัวอย่างอื่นๆ) โดยมีการก่อตัวของไพรเมอร์ไดเมอร์เป็นสัดส่วนผกผันกับความเข้มข้นของเทมเพลตอินพุต

รูปที่ 11 16 องศาเซลเซียสตัวอย่างของการใช้การวิเคราะห์เส้นโค้งละลายเพื่อระบุการปนเปื้อนของปฏิกิริยาใน NTC
รูปที่ 11 16 มิติระบุแอมพลิฟายเออร์ขนาดใหญ่ที่เป็นผลมาจาก PCR ของ gDNA

รูปที่ 11 17 A)โปรไฟล์ละลายและ B) การวิเคราะห์เจล agarose ของปฏิกิริยา SYBR GREEN I แม้ว่าโปรไฟล์การละลายจะแนะนำผลิตภัณฑ์ของ TM ที่แตกต่างกันแต่ภาพเจลแสดงว่ามีแอมพลิฟายเออร์เพียงตัวเดียว ซึ่งเป็นการบ่งชี้ลำดับแอมพลิฟายเออร์ที่มีบริเวณที่อุดมไปด้วย AT หรือ GC หรือองค์ประกอบที่ซ้ำกันทำให้เกิดการละลายที่ผิดปกติ
การเจือจางแบบอนุกรมของแม่แบบ/เส้นโค้งมาตรฐาน
ไม่ว่าการออกแบบทดลองจะมีข้อกำหนดสำหรับเส้นโค้งมาตรฐานสำหรับการวัดปริมาณในท้ายที่สุดหรือไม่การตรวจจับการเจือจางแบบอนุกรมของแม่แบบที่เหมาะสมเป็นวิธีการที่มีประสิทธิภาพในการตรวจสอบและแก้ไขปัญหาการวิเคราะห์ การตรวจจับการเจือจางแบบอนุกรมช่วยให้สามารถกำหนดช่วงไดนามิกเชิงเส้นในการทดสอบได้ รูปที่ 11.18 แสดงเส้นโค้งมาตรฐานที่มีจุดข้อมูลความเข้มข้นต่ำที่ไม่พอดีกับเส้นขอบแบบเส้นตรง เหตุผลที่พบบ่อยที่สุดสำหรับรูปแบบข้อมูลนี้คือไพรเมอร์ไดเมอร์เกิดขึ้นในตัวอย่างที่มีความเข้มข้นต่ำ (ดังแสดงใน รูปที่ 11 18 บี) นี่คือเส้นโค้งมาตรฐานที่สร้างขึ้นจากข้อมูลที่แสดงใน รูปที่ 11.2 รูปที่ 11.18 แสดงเส้นโค้งมาตรฐานที่มีตัวอย่างที่มีความเข้มข้นสูงหลุดออกมาจากช่วงเส้นตรง เหตุผลที่พบบ่อยที่สุดสำหรับเรื่องนี้คือการยับยั้งการตอบสนองหรือการตั้งค่าพื้นฐานไม่เหมาะสม

รูปที่ 11 18 a) จุดข้อมูลที่เกี่ยวข้องกับความเข้มข้นต่ำของเป้าหมายไม่ได้อยู่บนเส้นโค้งมาตรฐาน ข) นี่เป็นเรื่องปกติของปฏิกิริยาที่ส่งผลให้ไพรเมอร์ลดลงตามที่แสดงไว้ ในกรณีนี้จะไม่มีการสังเกตพบการเพิ่มขึ้นของ Cq สำหรับตัวอย่างที่มีความเข้มข้นต่ำ

รูปที่ 11 18 องศาเซลเซียสตัวอย่างที่มีเทมเพลตความเข้มข้นสูงจะไม่อยู่บนเส้นโค้งมาตรฐาน นี่เป็นปฏิกิริยาทั่วไปที่ถูกยับยั้งโดยความเข้มข้นของแม่แบบหรือเนื่องจากการตั้งค่าพื้นฐานที่ไม่ถูกต้อง
เส้นโค้งมาตรฐานยังใช้ในการวัดประสิทธิภาพของปฏิกิริยาในช่วงไดนามิกของการเจือจาง ต้องใช้ความระมัดระวังเพื่อให้แน่ใจว่าทุกจุดที่ใช้ในการคำนวณประสิทธิภาพอยู่ในสายการผลิต ปฏิกิริยาควรมีประสิทธิภาพใกล้เคียงกับ 100 เปอร์เซ็นต์มากที่สุดและผู้ที่มีประสิทธิภาพสูง (> 110%) หรือต่ำ (<85%) จะได้รับการตรวจสอบเพิ่มเติม

รูปที่ 11 19 กกรดนิวคลีอิกเทมเพลตถูกเจือจางผ่านชุด 10 เท่า แปลงขยายมีการไล่ระดับสีตื้นผิดปกติและ ΔCq เป็น 4 รอบมากกว่าที่คาดไว้ 3.3

รูปที่ 11 19 บาทการไล่ระดับสีของเส้นโค้งมาตรฐานของ Cq เทียบกับปริมาณจะใช้ในการคำนวณประสิทธิภาพของปฏิกิริยา

รูปที่ 11 20 กกรดนิวคลีอิกเทมเพลตถูกเจือจางผ่านชุด 10 เท่า ΔCq ระหว่างแปลงขยายคือ 1.5 รอบแทนที่จะเป็น 3.3

รูปที่ 11 20 บาทการไล่ระดับสีของเส้นโค้งมาตรฐานของ Cq เทียบกับปริมาณจะใช้ในการคำนวณประสิทธิภาพของปฏิกิริยาที่ใกล้เคียงกับ 140%

รูปที่ 11 20 องศาเซลเซียสการตรวจสอบโปรไฟล์โค้งละลายจะแสดงให้เห็นว่าตัวอย่างของความเข้มข้นที่ต่ำกว่า (ร่องรอยสีเหลืองและสีน้ำเงิน) ยังมีสัญญาณจากไพรเมอร์ที่ขยาย (สูงสุดที่ TM ที่ต่ำกว่า)
การแก้ไขปัญหากรณีศึกษา RT-PCR
การวิเคราะห์โพรบที่ล้มเหลว
การวิเคราะห์โดยใช้โพรบได้รับการออกแบบมาเพื่อตรวจจับ EIFB1 ในตัวอย่าง cDNA ของมนุษย์แต่ไม่มีการขยาย ปฏิกิริยาเริ่มต้นถูกรันบนเครื่องมือ ABI StepOne โดยใช้น้ำยาที่เข้ากันได้ มีความพยายามที่จะเพิ่มประสิทธิภาพของไพรเมอร์โดยใช้ช่วงของความเข้มข้นจาก 200 นิวตันเมตรถึง 900 นิวตันเมตร (รูปที่ 11.21) แต่ไม่มีการปรับปรุง การออกแบบการวิเคราะห์ได้รับการตรวจสอบและพบว่าเหมาะสมกับเป้าหมายและในซิลิโคคาดว่านี่เป็นการวิเคราะห์ที่มีคุณภาพสูง ไพรเมอร์ใหม่ถูกสังเคราะห์และทำงานควบคู่ไปกับอะลิควอทของการสังเคราะห์เดิมโดยใช้ตัวดำเนินการที่แตกต่างกัน SYBR Green I reagents (ดังนั้นตัวทำปฏิกิริยาที่แตกต่างกัน) และเครื่องมือ (Eppendorf Realplex) (รูปที่ 11.22) ในขณะที่ใช้วิธีการนี้เราตระหนักดีว่าวัตถุประสงค์หลักคือการแก้ปัญหาในขณะที่วัตถุประสงค์รองคือการอธิบายความล้มเหลว ปฏิกิริยานี้ทำให้เกิดการขยายตัวเทียบเท่าจากทั้งสองกลุ่มของไพรเมอร์ ในขั้นตอนนี้ปรากฏว่าปัญหาปฏิกิริยาวางอยู่กับโพรบและดังนั้นโพรบใหม่จึงถูกสังเคราะห์และทั้งสองชุดถูกเปรียบเทียบโดยผู้ประกอบการรายที่สองบนเครื่องมือ Realplex โดยใช้ รีเอเจนต์ LuminoCt ® (รีเอเจนต์ที่แตกต่างกันกับผู้ที่พยายามครั้งแรก) (รูปที่ 11.23) โพรบทั้งสองให้ข้อมูลการขยายตัวโพรบใหม่ปรากฏขึ้นดีกว่าโพรบเดิมเล็กน้อยแม้ว่าจะเป็นที่น่าสังเกตว่าโพรบเดิมถูกโพสต์ระหว่างห้องปฏิบัติการทดสอบและดังนั้นจึงอยู่ที่อุณหภูมิห้องในการแก้ปัญหาเป็นเวลาหลายวัน ในขั้นตอนนี้เป็นที่ชัดเจนว่าทั้งการวิเคราะห์เดิมและการทดแทนทำงานเมื่อดำเนินการโดยผู้ประกอบการรายที่สองใน รีเอเจนต์ LuminoCt ® บนอุปกรณ์ Realplex
ดังนั้นเหตุผลที่เหลือของความล้มเหลวเดิมจึงถือว่าเป็น:
- ผู้ประกอบการ: การทดลองซ้ำหลายครั้งโดยนักวิทยาศาสตร์ที่มีประสบการณ์และดังนั้นจึงถือว่าเป็นคำอธิบายที่ไม่น่าเป็นไปได้
- เครื่องมือ: อาจมีปัญหาบางอย่างเนื่องจากการวิเคราะห์อื่นๆล้มเหลว
- น้ำยา: คำอธิบายที่ง่ายที่สุดในการทดสอบ ตัว ทำปฏิกิริยา LuminoCt ® ถูกเปรียบเทียบกับตัวทำปฏิกิริยาที่มีอยู่โดยใช้ทั้งชุด oligo บนเครื่องมือ ABI StepOne โดยผู้ดำเนินการรายแรก ปฏิกิริยาล้มเหลวกับน้ำยาเดิมแต่ให้การขยายที่ดีกับ รีเอเจนต์ LuminoCt ® (รูปที่ 11.24)

รูปที่ 11 21ไพรเมอร์ถึง EIFB1 ได้รับการทดสอบที่ระดับความเข้มข้นระหว่าง 200 นิวตันเมตรและ 900 นิวตันเมตร ไม่พบการขยายสัญญาณภายใต้เงื่อนไขใดๆ (oligos และรีเอเจนต์ ABI ใน ABI StepOne Plus)

รูปที่ 11 22ไพรเมอร์สองชุดสำหรับ EIFB1 ถูกเปรียบเทียบใน SYBR Green I reagent; ชุดที่ล้มเหลวเดิมและชุดใหม่ (oligos และ reagents บนตราสาร Ependorf Realplex) ทั้งสองชุดของไพรเมอร์สนับสนุนการขยาย

รูปที่ 11 23มีการเปรียบเทียบไพรเมอร์และโพรบสองชุดกับ EIFB1 ในรีเอเจนต์ LuminoCt ® ชุดที่ล้มเหลวเดิมและชุดใหม่ (oligos และ reagents บนตราสาร Ependorf Realplex) ทั้งสองชุดของ oligos สนับสนุนการขยาย

รูปที่ 11 24การทดสอบไพรเมอร์และโพรบ EIFB1 ทำงานในน้ำยาที่แตกต่างกันสองชนิด (รีเอเจนต์ ABI หรือ LuminoCt ® เดิม) ได้รับข้อมูลจากการวิเคราะห์นี้เมื่อรันโดยใช้รีเอเจนต์ LuminoCt ® เท่านั้น
ประสิทธิภาพการตอบสนองไม่ถูกต้องและแปรผันได้
การทดสอบดำเนินการในการเจือจางแบบอนุกรมของ oligo เทียมโดยใช้ไพรเมอร์มาตรฐานและการทดสอบโพรบ การทดสอบได้รับการพัฒนาและปรับให้เหมาะสมกับเครื่องมือที่แตกต่างกันแต่ผลการเจือจางแปลกไม่ได้คาดว่าเมื่อถ่ายโอนไปยังห้องปฏิบัติการทดสอบและเครื่องมืออื่น (รูปที่ 11 25 เอ) เงื่อนไขการวิเคราะห์ทั้งหมดได้รับการปรับให้เหมาะสมอีกครั้งเพื่อให้เฉพาะเจาะจงกับห้องปฏิบัติการใหม่แต่ไม่มีการเปลี่ยนแปลงข้อมูล ผู้ปฏิบัติงานสังเกตว่าผลที่ได้จะเด่นชัดขึ้นเมื่อทำการวิเคราะห์ซ้ำภายในไม่กี่ชั่วโมงโดยใช้ชุดการเจือจางเดียวกัน ในฐานะที่เป็นส่วนหนึ่งของกระบวนการแก้ไขปัญหาการวิเคราะห์ถูกรันบนเครื่องมืออื่นโดยผู้ปฏิบัติงานคนอื่นที่สร้างเส้นโค้งมาตรฐานที่คาดไว้อีกครั้ง ซึ่งจะนำไปสู่ข้อเสนอแนะว่าปัญหาเบื้องต้นเกิดจากข้อผิดพลาดของผู้ปฏิบัติงานความล้มเหลวของเครื่องมือหรือความแปรผันเล็กน้อยในขั้นตอนการทดลอง เนื่องจากผู้ปฏิบัติงานทั้งสองมีประสบการณ์สูงและเครื่องมือทำงานได้ดีสำหรับการทดลองอื่นๆจึงมีการตรวจสอบตัวเลือกความแตกต่างเพียงเล็กน้อย เบาะแสที่สำคัญคือการสังเกตความแปรปรวนของข้อมูลจากชุดการเจือจางเดียวกันหลังจากเก็บตัวอย่างที่อุณหภูมิ 4 25°C (รูปที่ 11.25 Aand 11B) ซึ่งจะนำไปสู่การตรวจสอบหลอดที่ใช้สำหรับชุดการเจือจางและการทดสอบทางเลือกต่างๆ หลังจากเปลี่ยนไปใช้หลอดปฏิกิริยา Ependorf ขนาด 1.5 มล. สำหรับชุดการเจือจางเส้นโค้งมาตรฐานที่คาดการณ์ไว้ถูกสร้างขึ้น (รูปที่ 11 25 องศาเซลเซียส) ซึ่งแสดงให้เห็นว่าจำเป็นต้องเลือกชีววิทยาโมเลกุลพลาสติกเก็บข้อมูลต่ำสำหรับ PCR และการวิเคราะห์เหล่านี้มีความไวต่อการเปลี่ยนแปลงที่ละเอียดอ่อนในโปรโตคอล

รูปที่ 11 25 กเทมเพลต oligo เทียมถูกเจือจาง 10 เท่าและตรวจพบโดยใช้การวิเคราะห์ที่ใช้โพรบเฉพาะ มีความแตกต่างที่ไม่สอดคล้องกันระหว่างแปลงขยาย

รูปที่ 11 25 บาทเทมเพลต oligo เทียมถูกเจือจาง 10 4 เท่า (นี่คือการเจือจางที่ตรวจพบในรูปที่ 11 A) และทิ้งไว้ที่ 25°C เป็นเวลาหลายชั่วโมงก่อนที่จะถูกตรวจพบโดยใช้การวิเคราะห์ที่ใช้โพรบเฉพาะ มีความแตกต่างที่ไม่สอดคล้องกันระหว่างแผนการขยายสัญญาณซึ่งรุนแรงขึ้นตามเวลาระหว่างการเจือจางและการทดสอบ

รูปที่ 11 25 องศาเซลเซียสเทมเพลต oligo เทียมถูกเจือจาง 10 เท่าลงในหลอดโมเลกุลชีววิทยาเกรดและตรวจพบโดยใช้การทดสอบที่ใช้โพรบเฉพาะ มีความแตกต่างที่สอดคล้องกันระหว่างแปลงขยายตามที่คาดไว้
สรุป—รายการตรวจสอบการแก้ไขปัญหา PCR
- ตรวจสอบคุณภาพของตัวอย่าง (วัสดุที่มีประสิทธิภาพลดลงจะทำให้เกิดผลลัพธ์ที่ผิดพลาด)
- ตรวจสอบโปรโตคอล RT เข้ากันได้กับการออกแบบ (เช่น Oligo-DT PRIMEDT ต้องมีการวิเคราะห์ qPCR ในลำดับที่ 3 ' 1 kb)
- ตรวจสอบการออกแบบการวิเคราะห์
- ตรวจสอบการควบคุมทั้งหมด
- ตรวจสอบไพรเมอร์โดยใช้ SYBR GREEN I ย้อม/ใช้เจล
- ตรวจสอบให้แน่ใจว่าการตั้งค่าซอฟต์แวร์ถูกต้อง (พื้นฐาน, การตรวจจับสีย้อม, ความเข้มข้นของมาตรฐาน)
- ตรวจสอบให้แน่ใจว่าความเข้มข้นของ ROX ใช้ได้กับเครื่องมือ (และไม่รบกวน multiplex)
- ตรวจสอบระดับการเรืองแสงของพื้นหลัง
- ตรวจสอบการติดฉลากโพรบด้วยการทดสอบ DNase I หรือการสังเคราะห์โพรบซ้ำ
เพื่ออ่านต่อ โปรดเข้าสู่ระบบหรือสร้างบัญชีใหม่
ยังไม่มีบัญชีใช่หรือไม่?