Protokół ekstrakcji policzkowego DNA i amplifikacji WGA
Ten protokół zapewnia prostą i wygodną metodę izolacji, amplifikacji i oczyszczania genomowego DNA z wymazów z policzka. Wymazy z policzka są wygodną metodą pozyskiwania próbki DNA. Po wyizolowaniu DNA za pomocą protokołu ekstrakcji miniprep, można go amplifikować za pomocą protokołu amplifikacji całego genomu.
Protokół miniprep genomowego DNA
W celu uzyskania najlepszych wyników, do tego procesu należy użyć zestawu GenElute Mammalian Genomic DNA Miniprep Kit (G1N10).
- Osusz zebrane wymazy w temperaturze pokojowej przez 15 minut.
- Dodaj 280 μL Roztworu do Lizy T i 20 μL Proteinazy K. Włóż wymazówkę i delikatnie odwiruj. Zamknąć probówkę i wymieszać przez worteksowanie.
- Ikubować próbkę w temperaturze 55°C przez 20 minut z okazjonalnym worteksowaniem.
- Dodać 200 μL Roztworu do Lizy C i dokładnie worteksować przez 15 sekund.
- Inkubować w temperaturze 70 °C przez 10 minut.
- Dodać 500 μL Roztworu Przygotowującego Kolumnę do każdej Kolumny Wiążącej GenElute Miniprep (czerwony o-ring) i odwirować przy 12 000 × g przez 1 minutę. Wyrzucić płyn przepływowy.
Uwaga: Roztwór przygotowujący kolumnę maksymalizuje wiązanie DNA z membraną, co skutkuje bardziej spójną wydajnością - Dodaj 200 μL 95-100% etanolu do lizatu z kroku 5.
- Dokładnie wymieszaj przez worteksowanie i dodaj całą zawartość probówki do kolumny wiążącej.
- Wiruj z prędkością ≥6500 × g przez 1 minutę.
- Odrzuć probówkę zbierającą zawierającą przepływ i umieść kolumnę wiążącą w nowej probówce zbierającej o pojemności 2 ml.
- Dodaj 500 μL roztworu płuczącego (pamiętaj o rozcieńczeniu etanolem przed pierwszym użyciem) i wiruj z prędkością ≥6500 × g przez 1 minutę.
- Odrzuć probówkę zbierającą i przepływową i umieść kolumnę wiążącą w nowej probówce zbierającej o pojemności 2 ml.
- Dodaj kolejne 500 μL roztworu płuczącego do kolumny wiążącej i wiruj z maksymalną prędkością (12 000-16 000 × g) przez 3 minuty, aby wysuszyć kolumnę wiążącą.
- Nanieść pipetą 200 μL roztworu do elucji na kolumnę wiążącą i wirować przez 1 minutę z prędkością ≥6500 × g.
- Przechowywać wymyte DNA w temperaturze -20 °C lub przejść do etapu amplifikacji.
Uwaga: W przypadku korzystania z WGA2 nie ma potrzeby dostarczania polimerazy DNA, ponieważ enzym jest dostarczany wraz z zestawem
Protokół amplifikacji całego genomu (WGA)
.Protokół amplifikacji całego genomu GenomePlex przeprowadzonej przy użyciu zestawu GenomePlex Whole Genome Amplification Kit (WGA1) i/lub zestawu Complete Whole Genome Amplification Kit (WGA2).
Fragmentacja
- Przygotuj roztwór DNA o stężeniu 1 ng/ml z protokołu ekstrakcji krwi pełnej opisanego powyżej.
- Dodaj 1 µL 10X buforu do fragmentacji do 10 µL DNA (1 ng/µL) w probówce PCR.
- Umieść probówkę w termocyklerze w temperaturze 95 °C na dokładnie 4 minuty. Uwaga, inkubacja jest wrażliwa na czas i wszelkie odchylenia mogą zmienić wyniki.
- Natychmiast schłodzić próbkę na lodzie i krótko odwirować.
Przygotowanie biblioteki
- Dodaj 2 µL 1x buforu do przygotowania biblioteki.
- Dodaj 1 µL roztworu stabilizującego bibliotekę.
- Dokładnie wymieszaj i umieść w termocyklerze w temperaturze 95 °C na 2 minuty.
- Schłodź próbkę na lodzie i krótko odwiruj.
- Dodaj 1 µL enzymu przygotowującego bibliotekę, dokładnie wymieszaj i krótko odwiruj.
- Umieść próbkę w termocyklerze i inkubuj w następujący sposób:
16 °C przez 20 minut
24 °C przez 20 minut
37 °C przez 20 minut.br> 75 °C przez 5 minut
4 °C hold - Wyjmij próbki z termocyklera i krótko odwiruj. Próbki mogą być amplifikowane natychmiast lub przechowywane w temperaturze - 20 °C do trzech dni.
Amplifikacja
- Dodaj następujące odczynniki do całej 15 µL reakcji:
7.5 µL 10x Amplification Master Mix
47,5 µL Nuclease Free Water
5,0 µL JumpStart Taq DNA Polymerase (12.5 jednostek) dla WGA1
-lub-
5.0 µL polimerazy DNA WGA dla WGA2 - Dokładnie wymieszaj, krótko odwiruj i rozpocznij termocykling.br>
- Po zakończeniu cyklu, utrzymuj reakcje w temperaturze 4 °C lub przechowuj w temperaturze -20 °C, aż będą gotowe do analizy lub oczyszczenia.
Reamplifikacja
- Dodaj 10 µL 1 ng/mL amplifikowanego DNA WGA do probówki PCR lub płytki wielodołkowej.
Uwaga: Konieczne jest oczyszczenie reakcji WGA w celu zmniejszenia możliwego błędu w reamplifikacji. Zalecamy użycie naszego zestawu GenElute™ PCR Clean-Up Kit (numer produktu NA1020) lub standardowych metod oczyszczania, które izolują jedno- i dwuniciowe DNA. - Utwórz mieszaninę amplifikacyjną. Dla każdej reakcji reamplifikacji dodaj następujące składniki do amplifikowanego DNA WGA (krok 1):
47,5 µL wody wolnej od nukleaz
7.5 µL 10X Amplification Master Mix
5 µL WGA DNA Polymerase - Wiruj dokładnie, odwiruj krótko i rozpocznij termocykling. Poniższy profil został zoptymalizowany dla termocyklera PE 9700 lub równoważnego:
Denaturacja początkowa 95 °C przez 3 minuty
Wykonaj 14 cykli w następujący sposób:
Denaturacja 94 °C przez 15 sekund
Anneal/Extend 65 °C przez 5 minut - Po zakończeniu cyklu, utrzymywać reakcje w temperaturze 4 °C lub przechowywać w temperaturze -20 °C, aż będą gotowe do analizy lub oczyszczania. Stabilność DNA WGA jest równoważna genomowemu DNA przechowywanemu w tych samych warunkach.
Oczyszczanie amplifikowanych próbek wymazów z jamy ustnej
Oczyszczanie amplifikowanych produktów wykonywane przy użyciu zestawu GenElute PCR Clean-Up Kit (NA1020)
- Włóż kolumnę wiążącą GenElute Miniprep (z niebieskim O-ringiem) do dostarczonej probówki zbiorczej, jeśli nie została jeszcze zmontowana. Dodaj 0,5 ml roztworu przygotowującego kolumnę do każdej kolumny miniprep i odwiruj przy 12 000 x g przez 30 sekund do 1 minuty. Odrzucić eluat.
Uwaga: Roztwór przygotowujący kolumnę maksymalizuje wiązanie DNA z membraną, co skutkuje bardziej spójną wydajnością. - Dodaj 5 objętości roztworu wiążącego do 1 objętości reakcji PCR i wymieszaj. Na przykład, dodaj 500 µL roztworu wiążącego do 100 µL reakcji PCR. Przenieś roztwór do kolumny wiążącej. Wirować kolumnę z maksymalną prędkością (12 000 do 16 000 x g) przez 1 minutę. Odrzuć eluat, ale zachowaj probówkę zbierającą.
- Włóż ponownie kolumnę wiążącą do probówki zbierającej. Nałożyć 0,5 ml rozcieńczonego roztworu płuczącego na kolumnę i wirować z maksymalną prędkością przez 1 minutę. Odrzucić eluat, ale zachować probówkę zbiorczą.
Uwaga: Przed pierwszym użyciem należy dodać etanol do koncentratu roztworu płuczącego. Patrz instrukcja przygotowania. - Włóż ponownie kolumnę do probówki zbiorczej. Odwiruj kolumnę z maksymalną prędkością przez 2 minuty, bez dodatkowego roztworu płuczącego, aby usunąć nadmiar etanolu. Odrzuć wszelkie pozostałości eluatu, a także probówkę zbierającą.
- Przenieś kolumnę do świeżej probówki zbierającej o pojemności 2 ml. Nałożyć 50 µL roztworu elucyjnego lub wody na środek każdej kolumny. Inkubować w temperaturze pokojowej przez 1 minutę.
Uwaga: Podczas elucji wodą należy upewnić się, że pH wody wynosi od 5,5 do 8,5. Elucję można również przeprowadzić przy użyciu roztworu elucyjnego rozcieńczonego 10-krotnie wodą. - Aby eluować DNA, odwiruj kolumnę z maksymalną prędkością przez 1 minutę. Produkt amplifikacji PCR jest teraz obecny w eluacie i jest gotowy do natychmiastowego użycia lub przechowywania w temperaturze -20 °C.
Kwantyfikacja amplifikowanych próbek wymazu z policzka
Ilość DNA amplifikowanego przy użyciu amplifikacji całego genomu można wykryć z lub bez oczyszczania. Aby uzyskać najwyższą jakość próbek DNA, zdecydowanie zaleca się oczyszczenie próbek po amplifikacji. Amplifikowane produkty mogą być mierzone za pomocą testu PicoGreen® dsDNA Quantitation Assay firmy Molecular Probes Inc (7589). Inną metodą wykrywania amplifikowanych produktów jest absorpcja spektrofotometryczna (OD260) na spektrofotometrze NanoDrop® . Urządzenie to może mierzyć absorbancję na 1 μL próbki w szerokim zakresie dynamicznym (2-3 700 ng/μL).
Dane aplikacyjne dla amplifikacji próbki wymazu z jamy ustnej
Amplifikacja całego genomu przeprowadzona na próbce wymazu z jamy ustnej
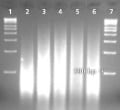
Zamplifikowane produkty są wizualizowane na 1,5% żelu agarozowym. Do każdej studzienki załadowano 5 µL amplifikowanego produktu. Amplifikowane produkty GenomePlex mają średni rozmiar 400 bp. Wzór rozmazu różni się w zależności od źródła, jak pokazano na żelu.
Linia 1 - drabina 1 kb
Linia 2 - krew
Linia 3 - roślina
Linia 4 - wymaz z policzka
Linia 5 - gleba
Linia 6 - kontrola pozytywna
Linia 7 - drabina 1 kb
Materiały
Response not successful: Received status code 500
Wymagane materiały dodatkowe
- Wymazówka z policzka
- 1.Probówki do mikrowirówki o pojemności 5 mL
- Mikrowirówka (z rotorem na probówki o pojemności 2 mL)
- Łaźnia wodna o temperaturze 55 °C lub blok cieplny
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?Dla wygody naszych klientów ta strona została przetłumaczona maszynowo. Dołożyliśmy starań, aby zapewnić dokładne tłumaczenie maszynowe. Tłumaczenie maszynowe nie jest jednak doskonałe. Jeśli tłumaczenie maszynowe nie spełnia Twoich oczekiwań, przejdź do wersji w języku angielskim.