Impatto del metodo di purificazione GenElute™-E sull'accuratezza della quantificazione del DNA e sulle procedure enzimatiche successive
Questi metodi che prevedono adsorbimento-lavaggio-eluizione sono ripetitivi e richiedono numerosi lavaggi e centrifugazioni. I molteplici passaggi in centrifuga possono causare un grado significativo di frammentazione del DNA. Infine, i sali caotropici e altri contaminanti possono facilmente essere trasferiti nel DNA o RNA eluito, compromettendone la purezza finale e la quantificazione, oltre che le procedure enzimatiche successive come la PCR.
Per questi motivi, abbiamo messo a punto e valutato un nuovo sistema di purificazione degli acidi nucleici che utilizza un’unica centrifugazione ed elimina le fasi di adsorbimento in alta concentrazione salina e di lavaggio con etanolo. I kit GenElute™-E per la purificazione di DNA e di RNA impiegano un metodo basato sulla cromatografia negativa che ricorre all'esclusione dimensionale per separare le grandi molecole di DNA e RNA dai composti proteici, lipidici e ionici più piccoli presenti nei campioni di cellule, tessuti, sangue e di altro tipo (Figure 1-2).
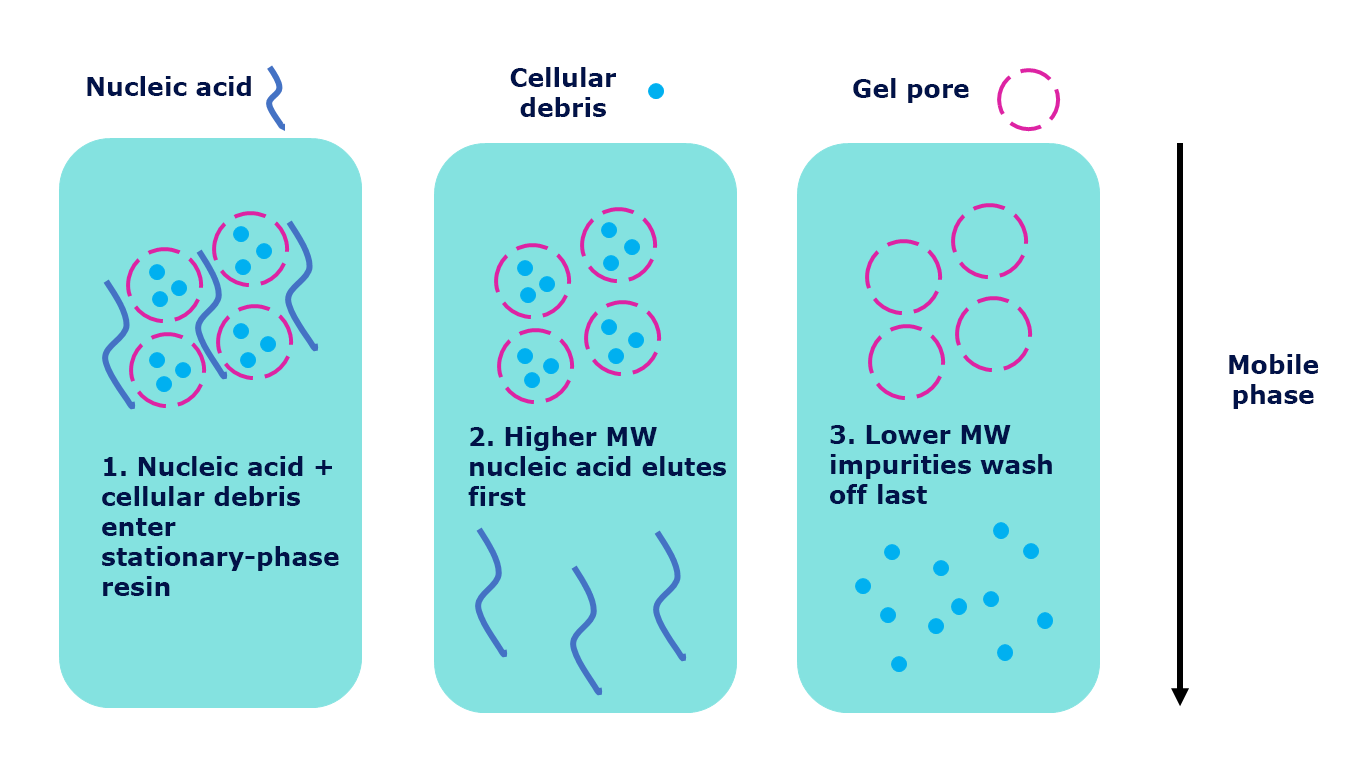
Figura 1.Purificazione degli acidi nucleici mediante la tecnica della cromatografia negativa (a esclusione dimensionale).
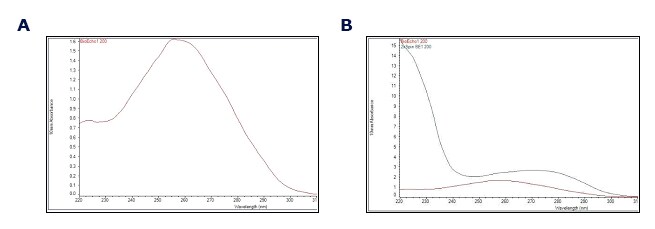
Figura 2.Eluizione di DNA purificato e ritenzione di proteine, lipidi e altre molecole utilizzando il kit GenElute™-E Single Spin Blood DNA High Yield (per la purificazione ad alta resa del DNA da sangue mediante un’unica centrifugazione) con campioni di sangue di topo. A) Spettro di assorbanza del DNA genomico purificato (linea rossa). B) Spettro di assorbanza del retentato della colonnina da centrifuga GenElute™-E (linea nera) sovrapposto allo spettro del DNA purificato (rosso). Gli spettri dei retentati mostrano le proteine e altri contaminanti con OD a ~280nm o ≤240nm. Si noti il cambio di scala sull’asse y.
Abbiamo stimato l’impatto del metodo di purificazione degli acidi nucleici sulle impurezze che interferiscono con la quantificazione e le procedure enzimatiche successive. Per questi studi comparativi, il DNA genomico è stato preparato impiegando la tecnologia di purificazione GenElute™-E che richiede un’unica centrifugazione oppure il classico metodo basato su silice del Fornitore X che prevede adsorbimento-lavaggio-eluizione. La purezza dell'acido nucleico è stata valutata con tre metodi:
- spettrofotometria UV (rapporto tra densità ottiche così misurato: A260nm/280nm e A260nm/230nm)
- elettroforesi su gel
- PCR quantitativa (qPCR)
I risultati indicano che con i sistemi per la purificazione di DNA e RNA GenElute™-E che richiedono un’unica centrifugazione si ottiene una purezza superiore rispetto alle consuete tecniche di preparazione basate sulla centrifugazione su silice, con una quantificazione più accurata e una minor interferenza dei contaminanti nelle procedure enzimatiche successive come la PCR.
Stima delle impurezze introdotte durante la preparazione degli acidi nucleici
Interferenza con la quantificazione effettuata mediante spettrofotometria UV
I comuni contaminanti biologici, come proteine, acidi nucleici frammentati, ssDNA, RNA (se si esamina il DNA), primer e sali caotropici derivati dai protocolli di purificazione ed estrazione, possono comportare una sovrastima della concentrazione di acido nucleico. Alcune di queste impurezze sono misurabili con la spettrofotometria UV, in cui un fascio di luce ultravioletta attraversa il campione di DNA o RNA purificato e l’assorbanza corrispondente viene misurata a diverse lunghezze d’onda.
L’assorbanza a 260 nm (A260) si usa per misurare gli acidi nucleici, mentre l’assorbanza a 280 nm (A280) è usata per misurare le proteine contaminanti del campione. Il rapporto A260/A280 permette di stimare la purezza del campione rispetto ai contaminanti proteici. Un DNA ad alta purezza avrà un rapporto A260/A280 ≥1,8. Rapporti più bassi indicano la presenza di contaminanti proteici nella preparazione finale.
L’assorbanza a 230 nm (A230) può essere usata anche per identificare la presenza di contaminanti chimici come i sali caotropici. Idealmente, il rapporto A260/A230 deve risultare maggiore di 2,0 per avere un’interferenza chimica minima nelle successive procedure enzimatiche come la PCR. Quando è necessario disporre di campioni ad alta purezza è fondamentale misurare questo rapporto.
Sono stati stimati lo spettro di assorbanza, il rapporto A260/280 e il rapporto A260/230 ottenuti con i sistemi per la purificazione di DNA e RNA GenElute™-E utilizzando dei campioni di sangue. I risultati sono stati confrontati con quelli ottenuti con un sistema di purificazione basato sulla centrifugazione su silice di un fornitore concorrente, utilizzando gli stessi campioni (Figura 3 e Tabella 1). I dati suggeriscono che con i sistemi di purificazione GenElute™-E si ottiene una maggiore purezza del DNA genomico, con meno contaminanti biologici e chimici nel campione finale rispetto ai campioni purificati con il classico metodo di adsorbimento su silice-lavaggio-eluizione.
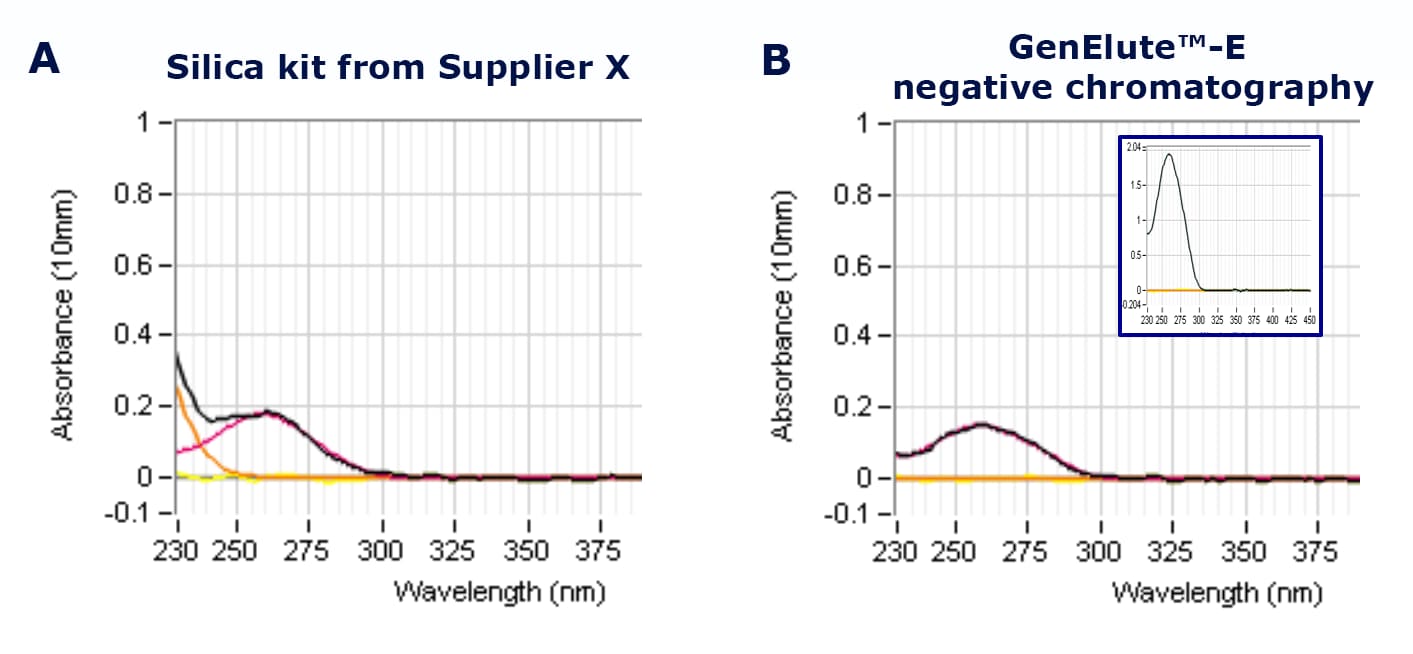
Figura 3.Spettro di assorbimento UV di DNA genomico preparato da campioni di sangue utilizzando A) il kit per la purificazione del DNA con centrifugazione su silice del Fornitore X o B) il kit GenElute™-E per la purificazione del DNA mediante un’unica centrifugazione e cromatografia negativa. Il grafico nel riquadro riporta la misurazione effettuata con la frazione eluita concentrata 10x. Le impurezze sono rilevate unicamente nelle frazioni eluite dalla silice. La linea nera corrisponde allo spettro del campione purificato con il metodo indicato e mostra anche l'apporto di tutti i contaminanti di processo. Lo spettro del campione purificato senza l'apporto dei contaminanti di processo, usato come controllo della linea di base, è indicato con la linea rossa. La linea arancione corrisponde allo spettro del metodo di purificazione in assenza di campione e mostra l’effetto dei contaminanti di processo sulla lettura spettrofotometrica.
| Tipo di campione | Metodo di purificazione | A260/280 (range ottimale: 1,8-2,0) | A260/230 (range ottimale: 2,0-2,2) |
|---|---|---|---|
| Sangue umano | Kit per la preparazione con centrifugazione su silice (Fornitore X) | 2,45 | 0,57 |
| Cromatografia negativa (a esclusione dimensionale) con GenElute™-E | 1,83 | 2,20 |
Stima mediante elettroforesi su gel del DNA frammentato e delle impurezze a basso peso molecolare
La separazione in base alle dimensioni ottenuta con l’elettroforesi su gel permette di valutare visivamente e quantitativamente la purezza del campione. Il DNA genomico di grandi dimensioni migra più lentamente rispetto ai frammenti di DNA e RNA più piccoli. Le bande risultanti possono essere poi osservate o quantificate con un colorante fluorescente.
I campioni di tessuto epatico di topo da cui è stato purificato il DNA mediante centrifugazione su silice (Fornitore X) o cromatografia negativa a esclusione dimensionale (kit GenElute™-E per la purificazione del DNA con un’unica centrifugazione) sono stati separati su un gel di agarosio. Come colorante per gli acidi nucleici è stato utilizzato il SYBR Green. Per normalizzare i risultati, in ciascuna corsia del gel è stata caricata la stessa massa di DNA di ciascun campione purificato. Visualizzando il gel di agarosio al transilluminatore è evidente come dalla cromatografia negativa (a esclusione dimensionale) si ottengano bande singole, ben più intense, rispetto a quelle ottenute con il metodo di purificazione basato su silice (Figure 4).
I campioni eluiti dopo centrifugazione su colonnine con membrana di silice presentano bande meno intense anche all’estremità inferiore del gel, indicative della presenza di molecole contaminanti più piccole costituite da RNA o DNA frammentato. Poiché il DNA dei campioni caricati sul gel era stato normalizzato (in base all’assorbanza a 260 nm), questi dati suggeriscono che tale metodo sovrastima la resa del DNA genomico perché anche le molecole contaminanti costituite da acidi nucleici più piccoli contribuiscono alla misurazione dell'assorbanza. Le singole bande, di maggiore intensità, del DNA ottenuto con il metodo della cromatografia negativa (a esclusione dimensionale) con GenElute™-E sono indice della maggior purezza del DNA genomico di questi campioni tissutali. I risultati sono stati quantificati con metodi fluorimetrici (Figura 5).
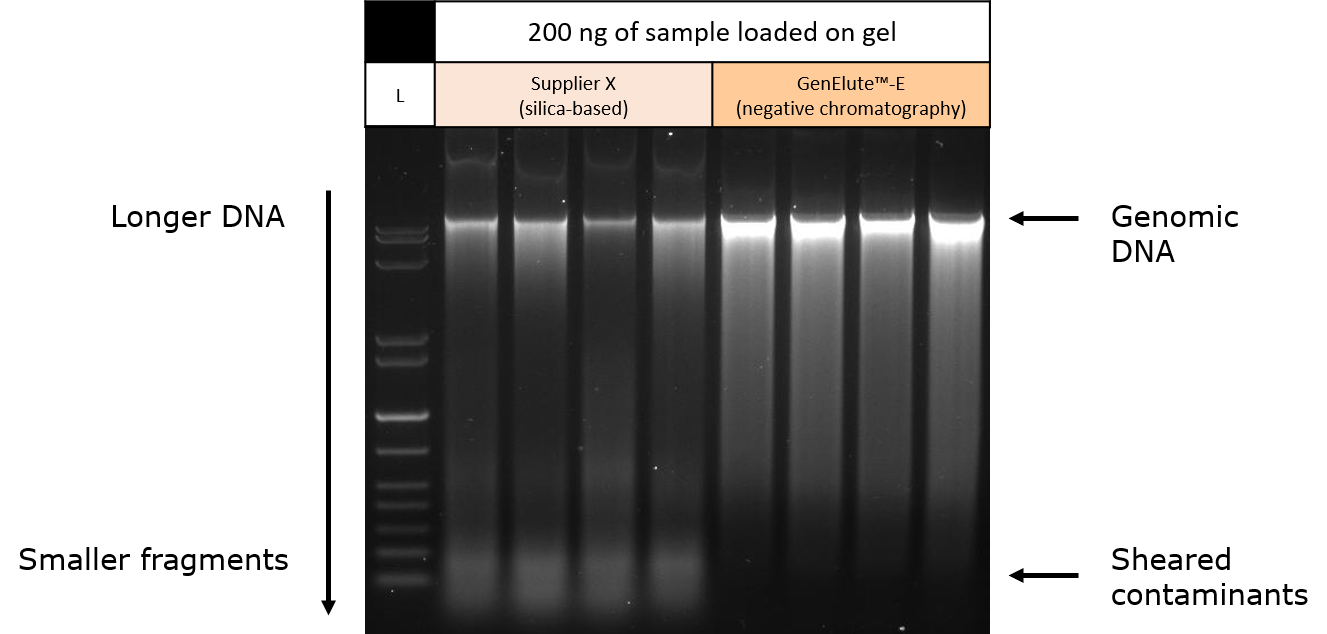
Figura 4.Elettroforesi su gel di DNA genomico purificato da campioni di tessuto epatico di topo. Il tessuto murino è stato purificato mediante centrifugazione su silice (Fornitore X) o cromatografia negativa a esclusione dimensionale (kit GenElute™-E per la purificazione del DNA con un’unica centrifugazione). Per caricare la stessa quantità di DNA in ogni corsia del gel di agarosio i campioni sono stati normalizzati. I risultati sono indicativi della presenza di RNA o DNA frammentato contaminante nei campioni purificati con il metodo su silice.
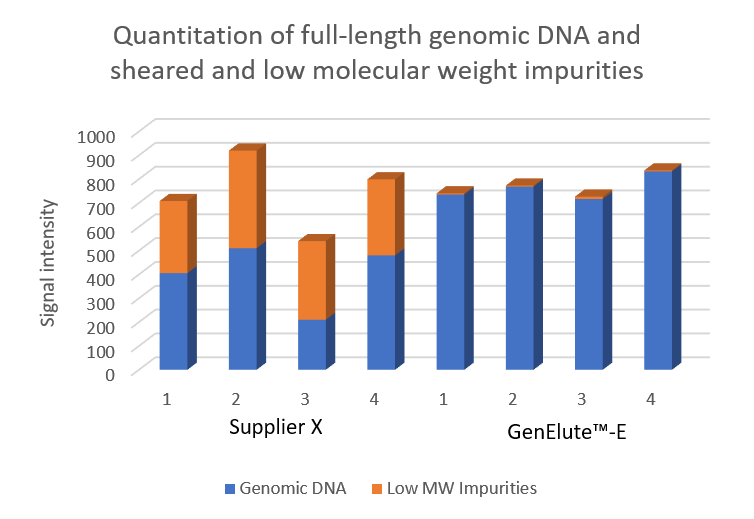
Figura 5.Resa calcolata del DNA genomico (gDNA) e quantità di RNA o DNA frammentato contaminante in campioni di tessuto epatico di topo. I campioni sono stati purificati mediante centrifugazione su silice (Fornitore X) o cromatografia negativa a esclusione dimensionale (kit GenElute™-E per la purificazione del DNA con un’unica centrifugazione). Il DNA è stato quantificato in base al segnale fluorescente emesso dal colorante STBR Green. Dai risultati emerge la presenza di quantità basse ma significative di RNA o DNA frammentato contaminante nei campioni purificati mediante centrifugazione su colonnine con membrana di silice.
Interferenza nella qPCR
I metodi di purificazione basati sulla silice introducono nel campione sali denaturanti e solventi organici, come l’etanolo, in fase di purificazione. È facile che questi contaminanti vengano trasferiti nelle applicazioni successive e che inibiscano le reazioni enzimatiche. La loro rimozione può rendere le procedure enzimatiche come la qPCR più sensibili e affidabili.
Per stimare la presenza di contaminanti potenziali inibitori dei processi enzimatici, in campioni di rene di topo è stato purificato il DNA genomico con i due metodi in oggetto. I campioni finali sono stati quindi impiegati per gli esperimenti di qPCR (Figura 6). Come controllo endogeno è stato amplificato anche il gene che codifica la beta-actina. Come si evince dal grafico, le curve di amplificazione dei campioni purificati su silice risultano spostate a destra rispetto a quelle dei campioni purificati con cromatografia negativa (a esclusione dimensionale) con GenElute™-E. Ciò suggerisce la presenza di contaminanti interferenti o una resa inferiore a quella calcolata nei campioni purificati su silice.
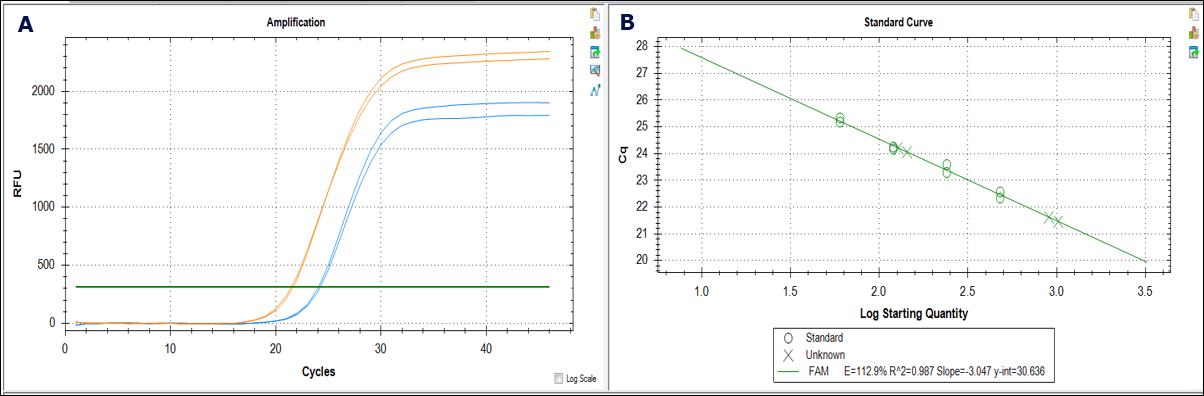
Figura 6.Analisi qPCR per la determinazione dei contaminanti interferenti in preparazioni di DNA genomico. A) Curve di amplificazione del DNA genomico da campioni di tessuto renale di topo purificato con un kit del Fornitore X che utilizza la centrifugazione su silice (curve in colore blu) o con il kit GenElute™-E (curve in colore arancione) che ricorre a un’unica centrifugazione e alla cromatografia negativa (a esclusione dimensionale). Le curve dei campioni purificati su silice risultano spostate a destra rispetto a quelle dei campioni purificati con GenElute™-E, a suggerire la presenza di contaminanti interferenti o una sovrastima della concentrazione iniziale nei campioni purificati su silice. B) Calcolo dei Cq utilizzando il gene della β-actina come controllo di riferimento endogeno.
Conclusioni
I kit GenElute™-E per la purificazione di DNA e RNA con un’unica centrifugazione forniscono un pratico metodo di cromatografia negativa per la purificazione degli acidi nucleici mediante esclusione dimensionale. Questi kit costituiscono un’alternativa alle tradizionali tecniche basate sulla centrifugazione su silice, perché migliorano la purezza dell’acido nucleico, come dimostrato dalla spettrofotometria UV, dall’elettroforesi su gel e dalla qPCR.
Materiali
Response not successful: Received status code 500
Per continuare a leggere, autenticati o crea un account.
Non hai un Account?