Polymerase-Kettenreaktion
Technischer Leitfaden für PCR-Technologien
Die Polymerase-Kettenreaktion (PCR)1,2,3 hat sich zu einer der am häufigsten verwendeten Techniken in der Molekularbiologie entwickelt. Sie wird in Anwendungen von der Grundlagenforschung bis zum Hochdurchsatz-Screening eingesetzt. Obwohl es sich um eine leistungsstarke Technik handelt, sind die allgemeine Akzeptanz und die vielfältigen Anwendungsmöglichkeiten auf ihre scheinbare Einfachheit und die relativ geringen Kosten zurückzuführen. Diese Technik wird zur Amplifikation spezifischer Ziel-DNA-Fragmente aus geringen Mengen Ausgangs-DNA oder -RNA verwendet (nach einem Schritt der reversen Transkription zur Herstellung komplementärer DNA (cDNA), siehe Reverse Transkription). Ein großer Vorteil der PCR besteht darin, dass Zielsequenzen aus einer einzigen Kopie des Ausgangsmaterials vervielfältigt werden können, selbst wenn das Template abgebaut und mit Inhibitoren kontaminiert ist. So wurden zum Beispiel Studien mit DNA aus altägyptischen Mumien durchgeführt, um die Mitglieder der Familie von König Tutanchamun zu identifizieren4. PCR taucht sogar in bekannten Filmen und in den meisten Krimiserien im Fernsehen in den Ermittlungen auf, auch wenn diese nicht ganz wirklichkeitsnah sind.
PCR-Zyklusverfahren
Eine typische PCR besteht aus:
- Anfänglicher Denaturierung: Die Reaktionstemperatur wird auf 95 °C erhöht und die Reaktion für 2-5 Minuten (bis zu 10 Minuten, abhängig von den Enzymeigenschaften und der Komplexität des Templates) inkubiert, um sicherzustellen, dass alle komplexen, doppelsträngigen DNA-Moleküle (dsDNA) für die Amplifikation in Einzelstränge aufgespalten werden.
- Zyklierung:
- Denaturierung: Die Reaktionstemperatur wird auf 95 °C erhöht, wodurch die gesamte dsDNA zu einzelsträngiger DNA (ssDNA) schmilzt (die Wasserstoffbrückenbindungen zwischen den komplementären Basen werden aufgebrochen).
- Hybridisierung: Die Temperatur wird auf etwa 5 °C unter die Schmelztemperatur (Tm) der Primer gesenkt (häufig 45-60 °C), um die Primerbindung an das Template zu fördern.
- Elongation: Die Temperatur wird auf den für die DNA-Polymerase-Aktivität optimalen Wert von 72 °C erhöht, damit die hybridisierten Primer verlängert (elongiert) werden können.
- Denaturierung: Die Reaktionstemperatur wird auf 95 °C erhöht, wodurch die gesamte dsDNA zu einzelsträngiger DNA (ssDNA) schmilzt (die Wasserstoffbrückenbindungen zwischen den komplementären Basen werden aufgebrochen).
- Wiederholung: Die Schritte 1-3 werden zyklisch durchgeführt, was zu einer exponentiellen Amplifikation des Amplikons führt (Abbildungen 2.1 und 2.2).
Die PCR besteht aus Zyklen, in denen die Reaktion erwärmt und abgekühlt wird. Jedes Temperaturplateau dient der Kontrolle eines bestimmten Reaktionsstadiums, und die Inkubationszeiten sind abhängig vom Gerät, den Reaktionsplatten oder -röhrchen und den Reagenzien. Diese sollen für jeden Versuch optimiert werden, insbesondere wenn eine höhere Nachweisempfindlichkeit erforderlich ist5.
Die anfängliche Denaturierungsphase besteht aus einem Zeitraum mit hoher Temperatur, in dem die Sekundärstruktur der komplexen doppelsträngigen DNA (dsDNA) zu einzelsträngiger DNA (ssDNA) geschmolzen wird. Da die DNA hohen Temperaturen ausgesetzt wird, muss diese Phase lang genug sein, um alle Stränge zu trennen, damit sie für das Priming zur Verfügung stehen. Aber nicht so lange, dass die DNA beschädigt wird. Eine Degradation während der ersten (oder nachfolgenden) Denaturierungsschritte oder eine unvollständige Denaturierung führt zu einer geringeren Nachweisempfindlichkeit5.
Während des kürzeren Denaturierungsschritts, der den Zyklus einleitet (10 Sekunden bis 1 Minute bei 95 °C), werden die DNA-Stränge der Zielsequenz getrennt, um, genau wie in der ersten Denaturierungsphase, Einzelstränge zu bilden. Die Reaktion wird dann auf die Primer-Hybridisierungstemperatur abgekühlt.
Der Hybridisierungsschritt (30 Sekunden bis 1 Minute bei Temperaturen von 45-60 °C) ist erforderlich, damit die Primer an die komplementäre Sequenz auf jedem der DNA-Einzelstränge binden. Die Primer sind so konzipiert, dass sie das Zielmolekül umklammern, und der dazwischen liegende Sequenzbereich wird als Amplikon bezeichnet. Im Allgemeinen kann davon ausgegangen werden, dass die Hybridisierungstemperatur 5 °C unter der Schmelztemperatur des Primer-Template-DNA-Duplex liegt.
Der letzte Schritt ist der Elongationsschritt (20 Sekunden bis 1 Minute bei 72 °C), der so durchgeführt wird, dass die DNA-Polymerase die Primersequenzen vom 3‘-Ende jedes Primers bis zum Ende des Amplikons verlängert. Eine 1-minütige Verlängerung reicht in der Regel aus, um PCR-Fragmente bis zu 2 Kilobasen (kb) zu synthetisieren. Um größere Fragmente zu amplifizieren, wird der Elongationsschritt auf 1 min pro kb verlängert. Während der ersten Verlängerung ist das Template nicht längenbegrenzt, sodass Templates synthetisiert werden, welche die Amplikonlänge überschreiten. In den nachfolgenden Verlängerungsschritten wird die Amplikonlänge durch die Primer-Sequenz an jedem Ende definiert.
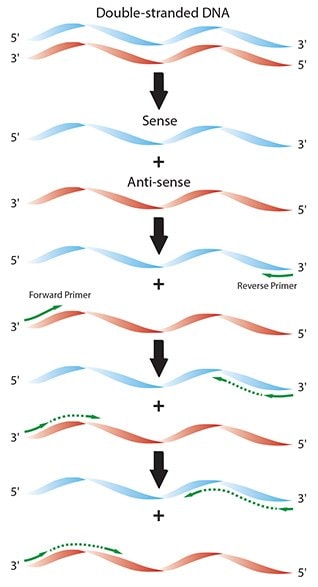
Abbildung 2.1.Schematische Darstellung der einzelnen Reaktionsabläufe in einer typischen PCR.

Abbildung 2.2.In einer theoretischen PCR verdoppelt sich die Menge des Zielamplikons mit jedem Zyklus, was zu einer exponentiellen Amplifikation der Zielsequenz führt (auf der X-Achse werden die PCR-Zyklen, auf der Y-Achse die Gesamtzahl der Amplikonmoleküle dargestellt).
Zu Beginn des zweiten Zyklus befinden sich zwei Formen des Templates in der Reaktion: die ursprünglichen DNA-Stränge und die neu synthetisierten DNA-Stränge, die aus der Primer-Sequenz, gefolgt von variablen Längen des am 3'-Ende verlängerten Amplikons, bestehen. Verbleibende ursprüngliche DNA-Stränge, die im ersten Zyklus nicht geprimt wurden, können im zweiten Zyklus eingefangen werden und resultieren in Molekülen, die aus Primer und Verlängerungsprodukt bestehen. Die Moleküle aus dem ersten Zyklus, die geprimt und verlängert wurden, dienen als Template für Primer, die komplementär zu dem neu synthetisierten Material sind.
Im dritten Zyklus besteht die neu synthetisierte DNA der Zielregion aus dem zweiten Zyklus nur noch aus dem Amplikon und wird somit zum spezifischen Template.
Der Zyklus wird kontinuierlich wiederholt, was zu einer exponentiellen Amplifikation der kopierten Sequenzen führt (Abbildung 2.2). Die Anzahl der erforderlichen Zyklen hängt von der gewünschten Ausbeute des PCR-Produkts ab. Diese hängt wiederum von der anfänglichen Kopienzahl und der Amplifikationseffizienz ab. Da die relative Konzentration vom Ausgangsmaterial zum Endprodukt unglaublich niedrig ist (z. B. 1/5,511, wenn ein einzelnes Molekül 40 Amplifikationszyklen durchläuft und bei jedem Zyklus eine Effizienz von 100 % erreicht wird, Abbildung 2.1), ist die am Ende der Reaktion entstehende DNA fast ausschließlich das PCR-Amplikon.
Obwohl die PCR theoretisch zu einer kontinuierlichen exponentiellen Amplifikation führen sollte, erreicht die Reaktion schließlich eine Plateauphase. Dies scheint auf eine unspezifische Bindung der DNA-Polymerase an DNA-Produkte zurückzuführen zu sein6,7. Dies wird bei der quantitativen Echtzeit-PCR (qPCR) und der Überwachung von Veränderungen der Fluoreszenzmarkierungen deutlich sichtbar (siehe Quantitative PCR).
Am Ende der Reaktion werden die Amplifikationsprodukte mittels Gelelektrophorese analysiert. Abhängig von der produzierten Menge und der Größe des amplifizierten Fragments können die Reaktionsprodukte direkt durch Färben des Gels mit Ethidiumbromid (Abbildung 2.3) oder mit neueren Farbstoffen wie BlueView™ 8 sichtbar gemacht werden.

Abbildung 2.3.Ein Beispiel für herkömmliche PCR-Produkte, die durch ein mit Ethidiumbromid gefärbtes Agarosegel aufgetrennt werden. Es wurde eine Duplex-PCR durchgeführt, um 2 Ziele, die sich in ihrer Größe unterscheiden, gleichzeitig nachzuweisen. Beide Fragmente sind als deutliche Banden zu erkennen (Bild mit freundlicher Genehmigung von Marion Grieβl).
PCR-Assay-Komponenten
Nachfolgend ist eine Zusammenfassung der Komponenten aufgeführt, die für die PCR erforderlich sind. Viele davon werden in speziellen Kapiteln sowie in den detaillierten Standard-PCR-Protokollen am Ende dieses Leitfadens (Anhang A) näher beschrieben.
Template
Es gibt verschiedene Methoden für die Aufreinigung von RNA und DNA. Diese sind zwar alle geeignet, aber mit großer Wahrscheinlichkeit funktionieren nicht alle gleich. Eine Kontamination des Templates mit anderen Materialien aus der Probenquelle kann zu einer Hemmung der PCR und im Extremfall zu einem vollständigen Ausfall des Assays führen. Es gibt einige Beispiele, die zeigen, dass Verunreinigungen sogar zu einer Verstärkung der Reaktion führen können9,10. Weitere Einzelheiten zur Template-Aufreinigung und Qualitätskontrolle finden Sie unter Probenaufreinigung und Qualitätsbewertung. Informationen zur reversen Transkription (RT) finden Sie unter Reverse Transkription. Üblicherweise werden 10 ng bis 1 μg genomische DNA (gDNA) zu 20-100 μl PCR-Assays hinzugefügt, während cDNA-Synthesereaktionen 1/5 bis 1/10 verdünnt und 5 μl zu 20-50 μl-Reaktionen hinzugefügt werden.
Primer
Oligonukleotid-Primer verfügen in der Regel über eine Länge von 15-25 Nukleotiden mit 40-60% GC und eine Tm, die für jedes Teil des Paars ähnlich ist. Eine einfache Methode zur Bewertung der Tm von kurzen Oligos (< 25 Basen) ist die Anwendung der Formel11:
Tm = 4(G + C) + 2(A + T)
Weitere Einzelheiten zum Primerdesign und zur Handhabung finden Sie unter Assay-Planung für PCR/qPCR/dPCR. Die ideale Ausgangstemperatur für die Hybridisierung liegt in der Regel 5 °C unter der Schmelztemperatur. Die Hybridisierungstemperatur kann mit einem Temperaturgradienten-PCR-Block optimiert werden. Ein Protokoll zur Temperaturoptimierung (am Beispiel der qPCR) finden Sie in Anhang A.
DNA-Polymerase
Es gibt eine Vielzahl von DNA-Polymerase-Enzymen und es ist entscheidend, dass das richtige Enzym anhand des Versuchsaufbaus ausgewählt wird (und nicht aufgrund der Verfügbarkeit eines bestimmten Enzyms im Gefrierschrank des Labors). Einige Enzyme sind beispielsweise so konzipiert, dass sie bei niedrigen Temperaturen inaktiv sind und in Hotstart-Protokollen verwendet werden, um eine unspezifische Amplifikation zu reduzieren, während andere Enzyme eine niedrige Fehlerrate aufweisen und für die Amplifikation von Fragmenten für die Klonierung ausgewählt werden.
dNTP
Die Konzentration der einzelnen dNTP (dATP, dCTP, dGTP, dTTP) in der PCR beträgt in der Regel 200 μM. Die dNTP-Konzentration muss jedoch im Überschuss vorhanden sein und für die Amplifikation von langen Fragmenten oder sehr häufig vorkommenden Zielen möglicherweise erhöht werden. In jüngster Zeit wurden die PCR-Pufferkomponenten verbessert, wie z. B. die CleanAmp™ dNTP12. Dabei handelt es sich um modifizierte Nukleosid-Triphosphate, die den Nukleotideinbau in die DNA-Polymerase blockieren. CleanAmp dNTP werden durch den anfänglichen Erhitzungsschritt und die nachfolgenden Denaturierungsschritte unter typischen Hot-Start-Zyklusbedingungen aktiviert. Durch dieses Verfahren wird die Menge der verfügbaren aktivierten dNTP während jedes PCR-Zyklus begrenzt, was eine spezifischere und effizientere Amplifikation des gewünschten Produkts ermöglicht. Sie reduzieren oder vermeiden vollständig ein Fehlpriming oder die Bildung von Primer-Dimeren und sind daher eine Alternative zu Hot-Start-DNA-Polymerasen.
MgCl2-Konzentration
Freie Mg2+ -Ionen sind als Cofaktor für die Aktivität der DNA-Polymerase erforderlich; Mg2+ -Ionen bilden jedoch Komplexe mit dNTP, Primern und DNA-Templates. Aus diesem Grund muss die optimale MgCl2-Konzentration für jeden Versuch ausgewählt werden, indem Reaktionen mit unterschiedlichen Konzentrationen getestet werden. Die MgCl2-Konzentration liegt in der Regel zwischen 1,5-5,5 mM, kann aber während der Optimierung im Bereich von 1-8 mM getestet werden (siehe Assay-Optimierung und Validierung).
Fluoreszierende Markierungen
Fluoreszenzfarbstoffe werden in die Reaktion integriert, wenn das Amplikon direkt nachgewiesen werden soll, z. B. bei Verwendung eines qPCR- oder digitalen PCR-Aufbaus (dPCR). Sie sind im Puffer enthalten oder an den Primern oder zusätzlichen Sonden angebracht (siehe Quantitative PCR und digitale PCR-Nachweismethoden).
Geräte
Ursprünglich war die Durchführung der PCR sehr arbeitsintensiv, da die Reaktionsröhrchen zwischen drei Wasserbädern hin- und herbewegt werden mussten, von denen jedes auf die erforderliche Temperatur für Denaturierung, Hybridisierung und Elongation eingestellt war. Zur Oberfläche der Reaktion wurde Öl hinzugefügt, um eine Verdunstung zu verhindern, und es war schwierig, die Analyse ohne Störungen durch das Öl durchzuführen. Heutzutage ist es üblich, einen der vielen spezialisierten Thermocycler zu verwenden, die im Handel erhältlich sind. Die Platten- und Blockformate variieren zwischen 48 , 96 oder 384 Wells und sind mit Mehrkanalpipetten kompatibel. In den meisten Fällen wird ein beheizbarer Deckel eingesetzt, durch den die Ölschicht überflüssig wird. Es gibt jedoch Hochdurchsatzgeräte, die auf dem ursprünglichen Wasserbadprinzip basieren13.
Anwendungsspezifische PCR-Modifikationen
Verschiedene Ableitungen der PCR erfordern Reaktionsänderungen. Einige dieser Modifikationen für gängige Anwendungen werden hier aufgeführt.
Klonierung
Die PCR kann zur Amplifikation ausgewählter Sequenzen verwendet werden, die in einen Vektor eingefügt werden sollen. Diese Sequenzen können so modifiziert werden, dass sie spezifische Regionen (Schwänze) für die Erkennung durch Klonierungsenzyme enthalten. Gegen den Vektor gerichtete Primer werden verwendet, um Fragmente zu isolieren, die bereits in Vektoren kloniert wurden. Für die PCR-Schritte ist ein DNA-Polymerase-Enzym mit niedriger Fehlerrate erforderlich.
Sequenzmarkierte Stellen
Diese sind in das 5'-Ende der Primer eingebaut und werden bei der Hochdurchsatz-Sequenzierung verwendet, wenn das spezifische Tag als Indikator dafür benötigt wird, dass ein bestimmtes Segment eines bestimmten Probengenoms in einem ausgewählten Klon vorhanden ist, sodass die Genomsequenzdaten dekonvolutiert und der ursprünglichen Ausgangsprobe zugeordnet werden können.
Ortsspezifische Mutagenese
Um die Funktion eines Proteins zu untersuchen, kann eine gewünschte Mutation in eine DNA-Sequenz eingefügt werden. Die gewünschte Basenänderung wird in die Primer (Richtung 5'-Ende) eingebaut, die auch die erforderlichen Restriktionsstellen für das Klonierungsenzym enthalten. Ein alternativer Ansatz ist ein mehrstufiges Protokoll, bei dem Primer, die für das Ziel spezifisch sind und die Klonierungsstellen enthalten, zusammen mit Primern verwendet werden, welche die gewünschte Mutation enthalten.
Materialien
Response not successful: Received status code 500
Literatur
Um weiterzulesen, melden Sie sich bitte an oder erstellen ein Konto.
Sie haben kein Konto?