Strategie di produzione per vaccini e terapie a base di mRNA
Con tempi ridotti dallo sviluppo alla clinica, la tecnologia dell'mRNA è promettente, non solo come agente rapido ed efficace per rispondere alle epidemie di malattie infettive, ma anche per lo sviluppo di nuove terapie per affrontare malattie con bisogni insoddisfatti. Le tecnologie a mRNA offrono molteplici vantaggi: un eccellente profilo di sicurezza, un elevato grado di versatilità e un processo di produzione templato.
Con il successo iniziale dei vaccini a mRNA, oggi i produttori di mRNA cercano di migliorare l'efficienza e la produttività dei loro processi di produzione migliorando la stabilità dell'mRNA e implementando strategie che migliorino la purificazione del pDNA e dell'mRNA e la scalabilità fino alla produzione GMP.
Visitate tutti i nostri prodotti e servizi per lo sviluppo e la produzione di mRNA.
Sezione Panoramica
Diverse decisioni chiave hanno un impatto enorme sul processo, sulla resa e sulla qualità del prodotto finale di mRNA, e una di queste è la qualità dei prodotti chimici di processo e delle materie prime. Soprattutto durante la trascrizione in-vitro e la purificazione a valle, l'mRNA non è protetto ed è ad alto rischio di degradazione enzimatica. L'uso di sostanze chimiche di alta qualità, testate per assenza di attività endonucleasica, riduce al minimo il rischio di degradazione indotta da RNasi e migliora la stabilità dell'mRNA durante la purificazione e la formulazione del prodotto farmaceutico a base di mRNA.
Questa pagina web vi aiuterà a superare queste e altre sfide, fornendovi informazioni sulle nostre soluzioni complete e integrate progettate per ottimizzare la produzione di mRNA. La nostra brochure "Process chemicals for mRNA drug manufacturing" fornisce i dettagli necessari per fare scelte informate.
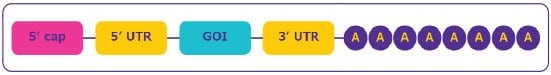
Figura 2.Struttura dell'mRNA.
- Regione del cappuccio all'estremità 5' della sequenza: Essenziale per la maturazione dell'mRNA, il riconoscimento da parte del ribosoma per una traduzione efficiente della proteina e la protezione dalla digestione delle nucleasi per una maggiore stabilità.
- Regioni non tradotte (UTR) nei domini a monte e a valle della regione codificante dell'mRNA: Regolano la traduzione, la localizzazione e la stabilità dell'mRNA; possono essere utilizzate per migliorare l'efficienza di espressione delle proteine.
- Open reading frame o regione della sequenza codificante: Contiene il gene di interesse (GOI).
- Coda poli(A): Cruciale per la traduzione delle proteine e la stabilità dell'mRNA, in quanto impedisce la digestione da parte dell'esonucleasi 3'.
Fare mRNA
La produzione di terapie e vaccini a base di mRNA inizia con un modello di DNA plasmidico (pDNA) che viene poi linearizzato e trascritto in mRNA.
- produzione di pDNA: Il modello di pDNA contiene un promotore di RNA polimerasi DNA-dipendente e la sequenza per il costrutto di mRNA. Il pDNA viene amplificato in cellule batteriche che vengono poi raccolte e lisate per rilasciare il pDNA circolare. Questo lisato è estremamente viscoso in quanto contiene il pDNA e altre impurità di grandi dimensioni e con carica negativa, come RNA, DNA genomico ed endotossine provenienti dalle cellule batteriche, il che rende difficile la purificazione.
- purificazione del pDNA: La purificazione del modello di pDNA dalle impurità deve massimizzare la separazione, riducendo al minimo il potenziale di danno meccanico al modello plasmidico. Il pDNA circolare purificato viene quindi linearizzato per evitare eventi di readthrough trascrizionale che potrebbero generare varianti indesiderate della sequenza di mRNA che dovrebbero essere rimosse.1 Questo modello di pDNA linearizzato viene ulteriormente purificato per rimuovere impurità come l'enzima di restrizione usato per la linearizzazione, l'albumina di siero bovino (BSA), frammenti di DNA ed endotossine. Poiché il modello di pDNA è tipicamente prodotto in cellule batteriche, le impurità da endotossine sono un'impurità critica che influisce sulle successive fasi di purificazione. Detergenti come Deviron® C16 detergent possono essere utilizzati per rimuovere efficacemente le endotossine e sono un'alternativa sostenibile, biodegradabile e conforme al REACH ai detergenti tradizionali.2
Ci sono notevoli differenze nell'approccio alla purificazione del pDNA su scala di laboratorio e di produzione GMP. I processi su scala di laboratorio utilizzano spesso tecniche di estrazione con solventi per separare il pDNA dagli altri componenti, ma la gestione e lo smaltimento di queste sostanze chimiche su larga scala possono essere impegnativi. Al contrario, gli ambienti di produzione GMP utilizzano tipicamente la filtrazione a flusso tangenziale (TFF) e la cromatografia per un'efficiente rimozione delle impurità.
Trascrizione in vitro (IVT): Il pDNA linearizzato e purificato viene quindi trascritto in mRNA in una reazione enzimatica.
- Componenti critici per la trascrizione in-vitro includono l'RNA polimerasi, che catalizza la trascrizione del DNA in mRNA; i ribonucleosidi trifosfati (rNTP), che servono come mattoni dell'mRNA; la pirofosfatasi inorganica (IPP), che aumenta la resa dell'mRNA; e gli inibitori della RNasi, che impediscono la degradazione dell'RNA. Il tampone di trascrizione contiene tipicamente ditiotreitolo (DTT) per ridurre i legami disolfuro e inibire l'attività della RNasi, mentre spermidina per migliorare l'efficienza della trascrizione e stabilizzare gli acidi nucleici. Per ridurre al minimo il rischio di degradazione enzimatica in questa fase critica del processo, è essenziale selezionare prodotti chimici testati per l'assenza di attività endonucleasica. Per una panoramica completa dei nostri prodotti chimici ed eccipienti di alta qualità, compresa un'ampia gamma di prodotti privi di endonucleasi, consultate la nostra brochure "Procedure chimiche per la produzione di farmaci a base di mRNA".
- Monitoraggio della trascrizione: I parametri critici di processo (CPP) devono essere monitorati durante la reazione IVT per controllare gli attributi critici di qualità (CQA) e assicurare un processo ottimale. Un monitoraggio efficace consente una produzione più controllata e risposte più rapide alla variabilità del processo.
Cap: Dopo la trascrizione, al trascritto di mRNA viene aggiunta una struttura di cap 5' per migliorare la stabilità e la trasduzione nella cellula ospite. Il cappuccio può essere aggiunto in due modi:
- Cappaggio co-trascrizionale: viene eseguito durante la fase IVT. Tuttavia, l'efficienza e la resa sono basse e questo approccio può generare impurità non capsulate a causa di un legame errato o di un'incorporazione inversa.
- Il capping enzimatico (o capping post-traslazionale) viene eseguito dopo la purificazione dell'mRNA. Questo approccio utilizza solitamente un enzima di capping per aggiungere il cappuccio all'mRNA purificato. Sebbene questo approccio sia più efficiente, è più costoso e rappresenta una fase di lavorazione aggiuntiva dopo la purificazione.
Purificazione dell'mRNA
In seguito alla trascrizione in-vitro trascrizione, l'mRNA viene purificato dalle impurità e dai materiali utilizzati nelle fasi precedenti, tra cui endotossine, RNA a doppio filamento (dsRNA) immunogeno, DNA template residuo, RNA polimerasi e impurità elementari. Sono disponibili diverse opzioni per purificazione dell'mRNA e rimozione del DNA residuo.
Separazione cromatografica come la coppia ionica in fase inversa (IPRP), scambio anionico (AEX) e la cromatografia di affinità (AC) con cattura di poli(dT) (Figura 3) purificano efficacemente l'mRNA target e rimuovono il DNA template, eliminando la necessità di digestione del DNA1, 3. La cromatografia viene utilizzata anche dopo il capping enzimatico per rimuovere trascritti di mRNA indesiderati e impurità oligonucleotidiche.

Figura 3:Confronto tra cromatografia in fase inversa a coppia ionica, a scambio anionico e di affinità per la purificazione di mRNA (DBC: capacità di legame dinamico).4,5
- L'accoppiamento ionico in fase inversa (IPRP) può essere utilizzato su piccola scala per catturare in modo efficiente l'RNA a singolo filamento (ssRNA) target e separarlo dalle impurità. Tuttavia, poiché questo metodo richiede solventi, è meno adatto per la produzione GMP ed è più indicato per la lucidatura che per la cattura.
- Cromatografia a scambio ionico (AEX) ha un'elevata capacità di legame dinamico e rimuove efficacemente impurità come dsRNA, RNA non tappato, ibridi RNA-DNA e altre strutture di RNA come l'mRNA a forcina. Sebbene l'AEX utilizzi soluzioni acquose, richiede agenti caotropici potenzialmente tossici e temperature operative fino a 85 °C per desorbire le molecole di mRNA di grandi dimensioni legate alla resina.
- Cromatografia di affinità (AC) per la cattura del poli(dT) utilizza una resina per catturare specificamente la coda di poli(A) dei trascritti di mRNA a lunghezza intera. Questo metodo rimuove efficacemente il DNA, i nucleotidi, gli enzimi, i componenti del tampone e qualsiasi altra impurità che non abbia una coda di poli(A). Per questo motivo, l'AC è comunemente usato per la cattura iniziale dell'mRNA, seguito dall'AEX per la lucidatura.
- La concentrazione finale e la diafiltrazione vengono eseguite dopo le fasi cromatografiche per massimizzare la purezza del prodotto e trasferire l'mRNA nel tampone appropriato per la formulazione o lo stoccaggio.
Considerazioni sullo scale-up
Le tecnologie monouso offrono scalabilità, adattabilità e qualità per i produttori con un'ampia pipeline di target e sono un fattore chiave per molti processi di produzione GMP di mRNA. La produzione GMP sfrutta i vantaggi delle fasi TFF o cromatografiche per una separazione efficiente su larga scala, sostituendo metodi di purificazione alternativi tipici dello sviluppo di processi su piccola scala. Le considerazioni da tenere a mente includono:
- Le fasi di TFF o cromatografia sostituiscono le fasi di estrazione con solvente e di precipitazione frequentemente utilizzate nello sviluppo del processo di mRNA.
- Ogni volta che è possibile, è necessario utilizzare sostanze chimiche di alta qualità e reagenti privi di endonucleasi per migliorare la stabilità dell'mRNA e ridurre al minimo la possibilità di degradazione dell'mRNA.
Negli ultimi anni si sono registrati importanti successi nell'introduzione di vaccini a base di mRNA su ampie popolazioni di pazienti. Sebbene le sfide rimangano, l'attenzione alla massimizzazione della stabilità dell'mRNA attraverso l'uso di sostanze chimiche di alta qualità e di reagenti privi di endonucleasi, l'ottimizzazione e l'adattamento delle tecnologie di purificazione cromatografica per massimizzare la separazione e semplificare lo scale-up, ha portato a importanti progressi nei modelli di produzione GMP.
Per garantire che la tecnologia dell'mRNA raggiunga il suo pieno potenziale, saranno necessarie soluzioni innovative, competenze e ingegno per assicurare l'evoluzione di piattaforme robuste su scala produttiva. Con i nostri prodotti, i nostri servizi e le nostre competenze tecniche, ci impegniamo a sviluppare soluzioni integrate per ottimizzare la produzione di mRNA.
Riferimenti
Per continuare a leggere, autenticati o crea un account.
Non hai un Account?Per comodità dei nostri clienti, questa pagina è stata tradotta da un sistema di traduzione automatica. Abbiamo fatto il possibile per fornire una traduzione automatica accurata, ma non possiamo garantire che essa sia perfetta. Se il contenuto della traduzione automatica non vi soddisfa, consultate la versione inglese.
